Innehållsförteckning:
- Författare John Day day@howwhatproduce.com.
- Public 2024-01-30 12:37.
- Senast ändrad 2025-01-23 15:10.
Av: Phuc Lam, Paul Yeung, Eric Reyes
Medveten om att fel i lungsegmentering kommer att ge falsk information om identifiering av ett sjukdomsområde och kan direkt påverka diagnosprocessen. Moderna datorstödstekniker lyckades inte ge exakta resultat när lungsjukdomar har utmanande former. Dessa onormala former kan orsakas av pleural effusioner, konsolideringar etc. Genom att använda lungsegmenteringstekniken, där lungans gränser isoleras från omgivande bröstvävnad, kan vår app identifiera gränserna med användarens inmatningströsklar för att ge helt anpassningsbara vyer av lungornas former, Syftet med detta MatLab-projekt är att skapa en användarvänlig interaktiv lungsegmenteringsapp för att upptäcka patologiska tillstånd i lungornas röntgenbilder. Vårt mål är att skapa ett mer effektivt sätt att illustrera och identifiera onormala lungor för att ge läkare och radiologer ett mer tillförlitligt sätt att diagnostisera lungsjukdomar. Med hjälp av appdesignverktyget i MatLab är programmet utformat för att fungera specifikt med bröströntgen och datortomografi (CT), men det testas också för att fungera med MR-skanningar.
Instruktionerna nedan innehåller vår brusfiltreringsteknik (lågpass Wiener-filter) samt bildtröskeln (med hjälp av intensitetshistogrammet för gråskala bilden) och med hjälp av en morfologisk gradient (skillnaden mellan vidgning och erosion av en bild) till identifiera en region av intresse. Instruktionen kommer sedan att förklara hur vi integrerar alla element i det grafiska användargränssnittet (GUI).
Notera:
1). Detta projekt är inspirerat av en forskningsartikel: "Segmentering och bildanalys av onormala lungor vid CT: aktuella tillvägagångssätt, utmaningar och framtida trender". Som finns här
2). Vi använder röntgenbilder från NIH: Clinical Center. Länk hittar du här
3). Hjälp för appdesigner hittar du här
4). Innan du kör koden: du måste ändra Dir -sökvägen (på rad 34) till din filkatalog och bildtyp (rad 35) (vi analyserar *.png).
Steg 1: Steg 1: Ladda bild
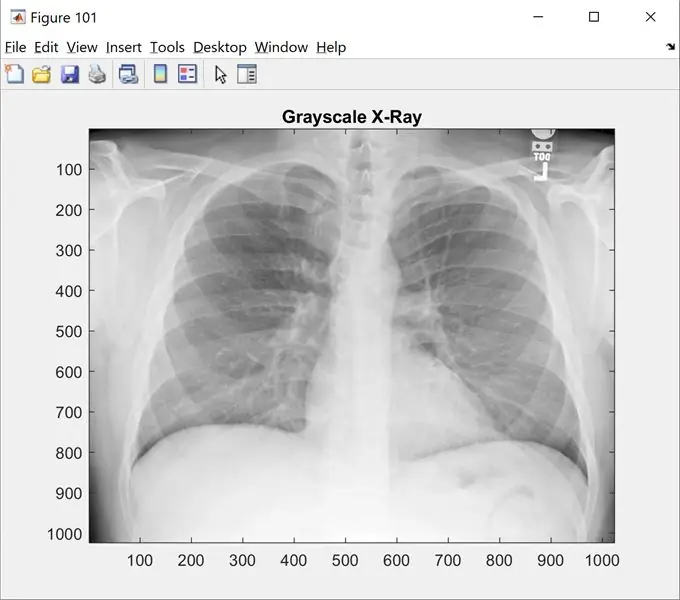
Detta steg visar den ursprungliga bilden i gråskala. Ändra 'name_of_picture.png' till ditt bildnamn
klar; clc; Stäng alla;
%% Läser in bilder
raw_x_ray = 'name_of_picture.png';
I = imread (raw_x_ray);
figur (101);
imshow (I);
färgbild (grå);
titel ('gråtonröntgen');
Steg 2: Steg 2: Brusfiltrering och histogram
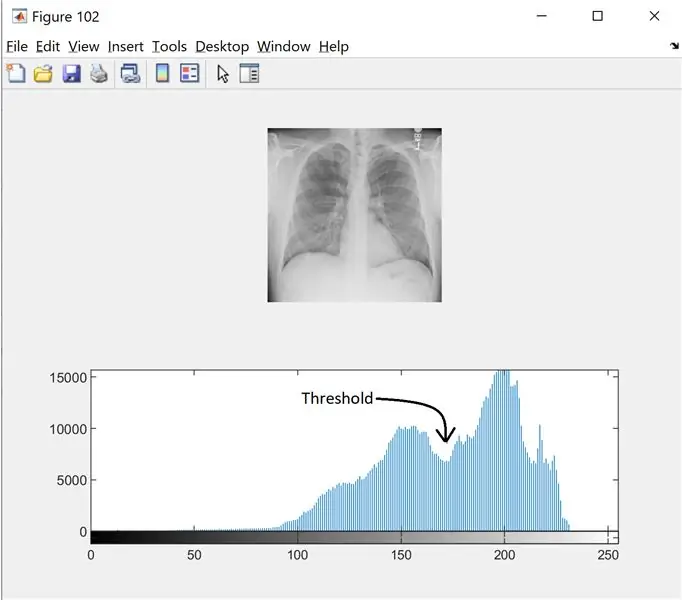
För att hitta tröskeln för gråskalebilden tittar vi på histogrammet för att se om ett distinkt läge. Läs mer här
I = wiener2 (I, [5 5]);
figur (102);
delplan (2, 1, 1);
imshow (I);
delplan (2, 1, 2);
imhist (I, 256);
Steg 3: Steg 3: Ställa in trösklar
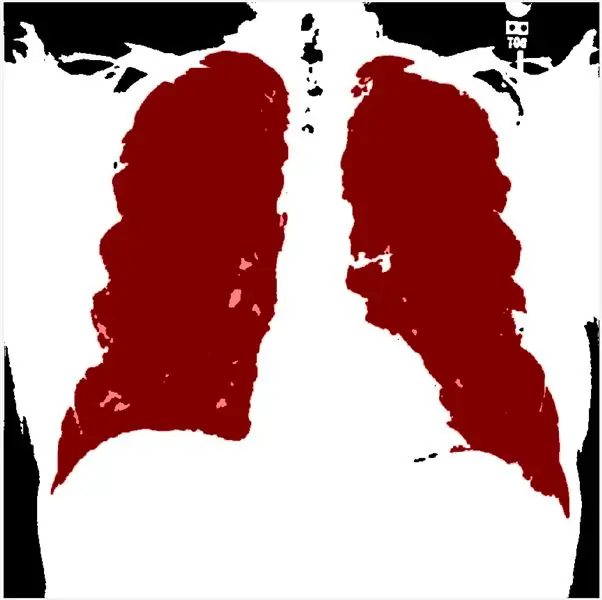
Detta steg låter dig ställa in tröskeln enligt histogrammet. morphologicalGradient kommer att markera området av intresse för rött, och funktionen gränsar överlagrar den skisserade och filtrerade bilden av lungan i rött.
Genom att använda regionprops kan vi exakta stabilitetsgrupperna och sortera dem i fallande. Därefter binariserar jag den grå sclae -bilden och tillämpar den morflogiska gradientmetoden och mLoren Shurasking för att markera regionen av intresse (ROI). Nästa steg är att vända bilden så att lungan ROI är vit i den svarta bakgrunden. Jag använder funktionen showMaskAsOverlay för att visa 2 mask. Obs: koden är inspirerad av Loren Shure, länk.
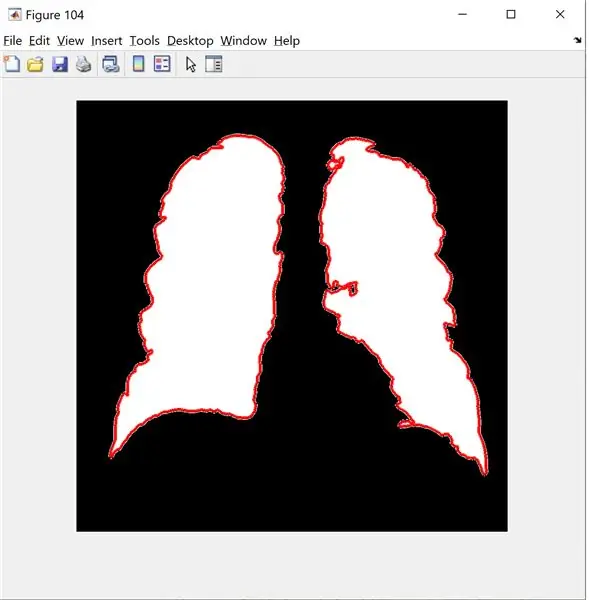
Lasly, jag skapar röd kontur genom att använda bwbwboundaries och maskera filterbilden och gränserna.
a_thresh = I> = 172; % anger denna tröskel
[labelImage, numberOfBlobs] = bwlabel (a_thresh);
rekvisita = regionprops (a_thresh, 'all');
sortedSolidity = sort ([props. Solidity], 'descend');
SB = sortedSolidity (1);
om SB == 1 % SB endast accepterar soliditet == 1 filtrera bort ben
binaryImage = imbinarize (I); figur (103);
imshow (binaryImage); färgbild (grå);
SE = strel ('square', 3);
morphologicalGradient = imsubtract (imdilate (binaryImage, SE), imerode (binaryImage, SE));
mask = imbinarize (morphologicalGradient, 0,03);
SE = strel ('square', 2);
mask = nära (mask, SE);
mask = imfill (mask, 'hål');
mask = bwareafilt (mask, 2); % kontrollantal områdeshow
notMask = ~ mask;
mask = mask | bwpropfilt (notMask, 'Area', [-Inf, 5000 - eps (5000)]);
showMaskAsOverlay (0,5, mask, 'r'); % du måste ladda ner app/funktion showMaskAsOverlay
BW2 = imfill (binaryImage, 'hål');
ny_bild = BW2;
new_image (~ mask) = 0; % invertera bakgrund och hål
B = bwboundaries (new_image); % kan bara acceptera 2 dimensioner
figur (104);
imshow (ny_bild);
vänta
gränsöverskridande (B);
slutet
Steg 4: Skapa GUI
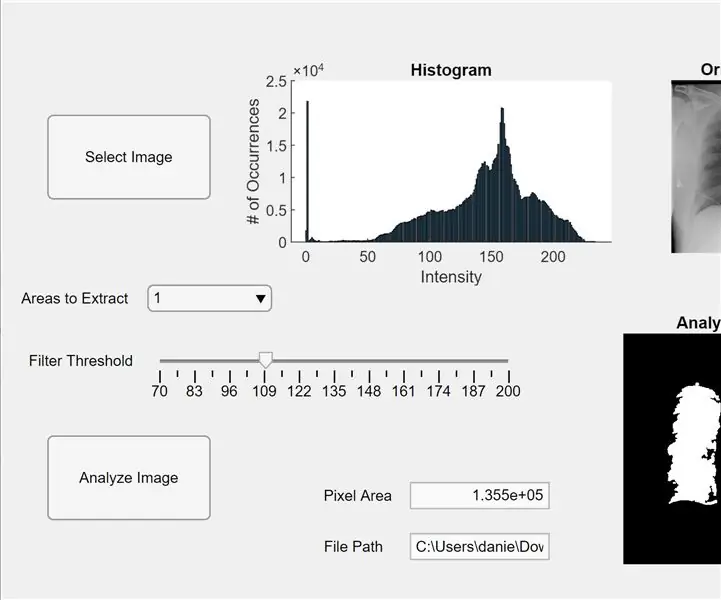
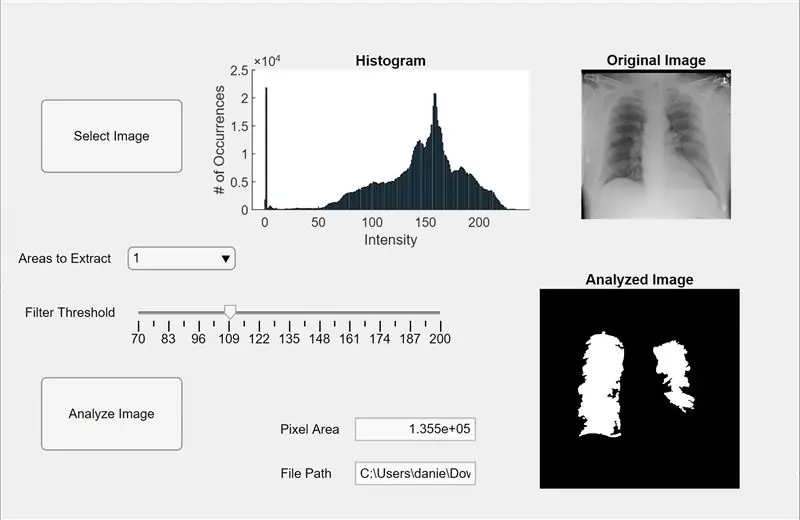
Nu integrerar vi den tidigare koden i en MATLAB -app. Öppna App Designer i MATLAB (Ny> App). Först designar vi gränssnittet genom att klicka-håll-och-dra i tre axlar in i mitten av arbetsytan. Därefter klickar och håller du ned två knappar, ett redigeringsfält (text), ett redigeringsfält (numeriskt), ett reglage och en rullgardinsmeny. Två axlar kommer var och en att visa förhandsgranskningen och analysera bilden, och den tredje axeln kommer att visa ett histogram med pixlar för förhandsgranskningen "vald" bild. Redigeringsfältet (text) -rutan visar filvägen för den valda bilden och redigeringsfältet (numeriskt) visar det identifierade pixelområdet för lungorna.
Byt nu från designvy till kodvy i App Designer. Ange koden för egenskaper genom att klicka på den röda knappen "Egenskaper" med ett plustecken. Initiera egenskaperna I, tröskel och regionToExtract som i koden nedan. Högerklicka sedan på en knapp i den övre högra sidan av arbetsytan (komponentbläddraren) och gå från Återuppringning> Gå till … återuppringning. Lägg till koden för “function SelectImageButtonPushed (app, händelse).” Denna kod låter dig välja en bild att analysera från din dator med uigetfile. Efter att ha valt en bild visas en förhandsgranskningsbild under axlarna tillsammans med ett histogram. Högerklicka sedan på den andra knappen och upprepa samma procedur för att skapa en återuppringningsfunktion.
Lägg till koden under "function AnalyzeImageButtonPushed (app, händelse)." Den här koden utför pixelräkningen och blobdetekteringen på förhandsgranskningsbilden på knappen analysera bild (beroende på vilken du högerklickade för den här koden). Efter programmering av knapparna programmerar vi nu reglaget och rullgardinsmenyn. Högerklicka på reglaget, skapa en återuppringningsfunktion och lägg till koden under "funktion FilterThresholdSliderValueChanged (app, händelse)" till slutet. Detta gör att reglaget kan justera tröskeln för grå intensitet.
Skapa en återuppringningsfunktion för rullgardinsmenyn och lägg till koden under "function AreastoExtractDropDownValueChanged (app, event)" för att låta rullgardinsmenyn ändra antalet blobbar som visas på de analyserade bildaxlarna. Klicka nu på varje enhet i komponentbläddraren och ändra deras egenskaper efter eget tycke, till exempel att ändra namn på enheter, ta bort axlar och ändra skalning. Dra och släpp enheterna i komponentbläddraren i designvyn till en funktionell och lättförståelig layout. Du har nu en app i MATLAB som kan analysera bilder av lungor för pixelområde!
fastigheter (Access = privat) I = ; % bildfil
tröskel = 257; %tröskel för binärisering av grå intensitet
regionToExtract = 2;
slutet
funktion SelectImageButtonPushed (app, händelse)
clc; Dir = 'C: / Users / danie / Downloads / images_004 / images'; %definiera invariat fil "prefix"
[imageExt, sökväg] = uigetfile ('*. png'); %ta tag i den variabla delen av bildnamnet
imageName = [Dir filesep imageExt]; %sammanfogade varierande och variabla strängar
app. I = imread (imageName); %läste bilden
imshow (app. I, 'parent', app. UIAxes); %visar bilden
app. FilePathEditField. Value = sökväg; %visningsfilväg för var originalbilden kom ifrån
slutet
function AnalyzeImageButtonPushed (app, händelse)
originalbild = app. I;
originalImage = wiener2 (app. I, [5 5]); %borttagningsfilter
histogram (app. AxesHistogram, app. I, 256); %visar histogram för bild
a_thresh = originalImage> = app.threshold; % anger denna tröskel
labelImage = bwlabel (a_thresh);
rekvisita = regionprops (a_thresh, 'all');
sortedSolidity = sort ([props. Solidity], 'descend');
SB = sortedSolidity (1);
om SB == 1 % SB endast accepterar soliditet == 1 filtrera bort ben
SE = strel ('square', 3);
morphologicalGradient = imsubtract (imdilate (labelImage, SE), imerode (labelImage, SE));
mask = imbinarize (morphologicalGradient, 0,03);
SE = strel ('square', 2);
mask = nära (mask, SE);
mask = imfill (mask, 'hål');
mask = bwareafilt (mask, app.regionsToExtract);
% kontrollantal områdeshow
notMask = ~ mask;
mask = mask | bwpropfilt (notMask, 'Area', [-Inf, 5000 - eps (5000)]);
BW2 = imfill (labelImage, 'hål');
ny_bild = BW2;
new_image (~ mask) = 0;
B = bwboundaries (new_image); % kan bara acceptera 2 dimensioner imshow (new_image, 'parent', app. UIAxes2);
håll (app. UIAxes2, 'on');
gränsöverskridande (B);
set (gca, 'YDir', 'reverse');
lungArea = bwarea (new_image);
app. PixelAreaEditField. Value = lungArea;
slutet
slutet
funktion FilterThresholdSliderValueChanged (app, händelse)
app.threshold = app. FilterThresholdSlider. Value;
slutet
function AreastoExtractDropDownValueChanged (app, event) stringNumber = app. AreastoExtractDropDown. Value;
app.regionsToExtract = str2double (stringNumber);
slutet
slutet
Rekommenderad:
MATLAB Enkel ansiktsigenkänning: 4 steg

MATLAB Enkel ansiktsigenkänning: Huvudsyftet med dessa instruktioner är att visa hur enkelt bildbehandlingen kommer att vara. Med hjälp av MATLABFace har detektering och spårning varit ett viktigt och aktivt forskningsfält, så det är därför jag ska förklara hur kan man göra det
Hur man kodar och publicerar Matlab 2016b till Word (nybörjarguide): 4 steg

Hur man kodar och publicerar Matlab 2016b till Word (nybörjarguide): Matlab är ett högpresterande språkprogram som används för att beräkna tekniska resultat. Den har förmågan att integrera bilder, beräkningar och programmering på ett användarvänligt sätt. Med det här programmet kan användaren publicera problem och lösningar
Kontrollera Raspberry Pi med Matlab: 5 steg

Kontrollera Raspberry Pi med Matlab: Hej, denna handledning handlar om att styra din huvudlösa hallon pi med matlab. Du kan behöva installera matlab till den senaste versionen för stöd för nyare hallon pi -kort
Matlab-baserad ROS robotstyrenhet: 9 steg
Matlab-baserad ROS Robotic Controller: Ända sedan jag var barn har jag alltid drömt om att vara Iron Man och gör det fortfarande. Iron Man är en av de karaktärer som är realistiskt möjligt och helt enkelt jag strävar efter att bli Iron Man någon gång även om folk skrattar åt mig eller säger att det är omöjligt
Förbättringar av träningsprogrammering i MATLAB: 6 steg

Tågprogrammeringsförbättringar i MATLAB: Mål: Målet med detta programmerade system är att titta på Arduino i liten skala och tillämpa kodningen i större skala för att potentiellt förbättra vissa säkerhetsfunktioner hos Amtrak Railroad -system. För att göra detta har vi lagt till en jordfuktighet sen
