Innehållsförteckning:
- Steg 1: Skapa Adjacency Matrix
- Steg 2: Skapa relationer
- Steg 3: Lägg till sjukdomsstatistik
- Steg 4: Slumpmässigt chansen att en vaccinerad och ovaccinerad person kan bli smittad
- Steg 5: Skapa matriser för människor som är ovaccinerade och infekterade från initial information
- Steg 6: Plotta inledande diagram
- Steg 7: Simulera utvecklingen av infektionen
- Steg 8: Använd Monte Carlo -teorin
- Steg 9: Gör filen ('infectSim.m') med simuleringen i en funktion
- Steg 10: Beräkna andelen ovaccinerade och vaccinerade som smittats
- Steg 11: Skapa en utmatningsvariabel i din funktion 'infectSim.m'
- Steg 12: Skapa en meny för att få de första villkoren för simuleringen från användaren
- Steg 13: Välj en procentandel av ovaccinerade personer och beräkna genomsnittet av ovaccinerade och infekterade för vald procentandel
- Steg 14: Graf: 'Trenden för infektion i ovaccinerade vs. Vaccinerad för specificerad sjukdom '
- Steg 15: Slutprodukt: Hur simuleringen ser ut
- Författare John Day day@howwhatproduce.com.
- Public 2024-01-30 12:46.
- Senast ändrad 2025-01-23 15:11.
Översikt över projektet:
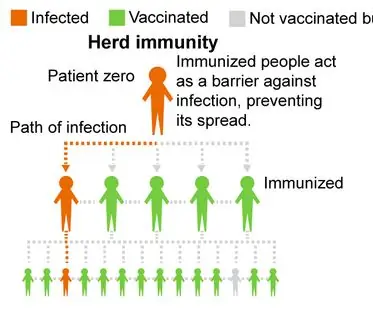
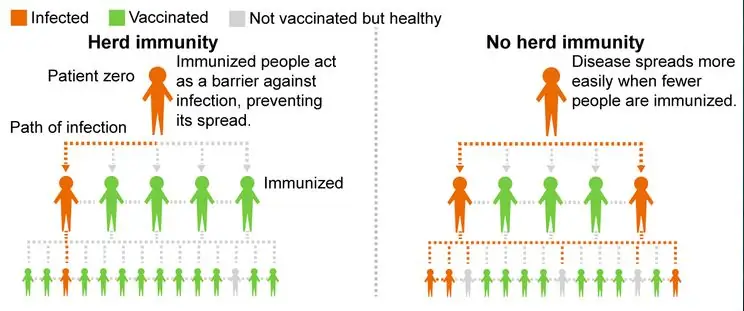
Vårt projekt utforskar flockimmunitet och hoppas kunna uppmuntra människor att vaccinera sig för att minska infektionsgraden i våra samhällen. Vårt program simulerar hur en sjukdom infekterar en befolkning med olika andelar av vaccinerade och ovaccinerade priser. Det visar besättningsimmunitet genom att visa hur ett ökat antal av den vaccinerade populationen kan minska antalet drabbade.
Vi modellerar detta i Matlab med hjälp av grafteorikoncept. Grafteori är ett matematiskt sätt att representera förhållandena mellan objekt. I grafteorin har grafer hörn (eller noder) anslutna med kanter (eller linjer). För vårt projekt är noderna de berörda individerna och kanterna är deras anslutningar. Till exempel, om två noder är anslutna till en kant betyder det att de är "vänner" eller har någon form av kontakt med varandra. Denna kontakt är ett sätt för sjukdomen att sprida sig. Det är därför vi använde grafteori för att modellera vårt koncept eftersom vi ville se hur sjukdomen sprider sig bland individer som är kopplade till en befolkning.
Vårt projekt omfattar också Monte Carlo -metoden. Monte Carlo -metoden är algoritmer som skapar upprepade slumpmässiga provtagningar för att få numeriska resultat. I vårt projekt använder vi denna metod för att köra vår simulering flera gånger och ändra andelen initiala ovaccinerade för att se hur snabbt människor blir smittade.
All projektkod är länkad längst ner!
PC-kredit:
Matlab-länk till Graph Theory:
Steg 1: Skapa Adjacency Matrix
Skapa ett nytt skript. Vi kommer att kalla vår infektion SimM.
Vi ska skapa en variabel 'NUMOFPEOPLE'. Du kan tilldela det till vilket heltal som helst. Detta kommer att representera antalet personer i din befolkning.
Från och med nu kommer vi att anta det
NUMOFPEOPLE = 20;
Börja först med att använda Matlabs grafteorifunktioner för en oreglerad graf.
Om du är intresserad av att lära dig mer, här är en länk där du kan läsa mer på djupet.
www.mathworks.com/help/matlab/math/directed-and-undirected-graphs.html
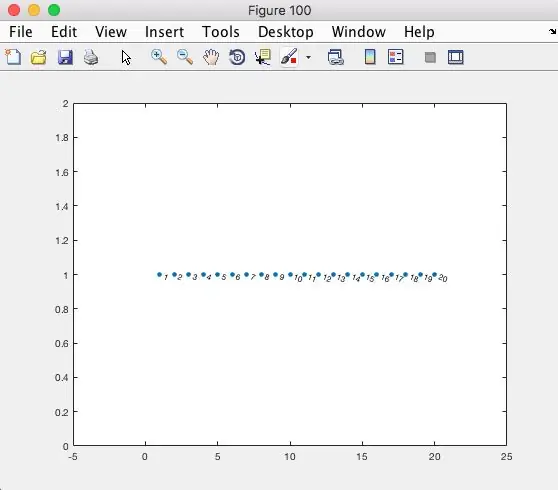
Skapade en adjacensmatris.
adjMatrix = nollor (NUMOFPEOPLE);
Detta skapar en kvadratisk matris på 0s. Varje rad i matrisen är en person. Varje kolumn i matrisen är en person eller vän som personen träffar under hela dagen.
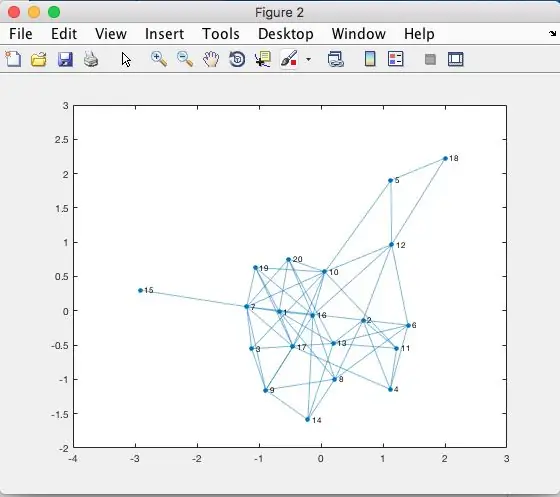
Se figur 100 (ovan) för att visualisera hur adjMatrix ser ut för 20 personer.
** Från och med nu antar vi att NUMOFPEOPLE är lika med 20. **
Du kan försöka rita denna adjacensmatris. Här är lite mer information om att plotta dessa typer av matriser.
Obs: Hur adjacensmatrisen fungerar.
Ex:
%gör den intilliggande matrisen
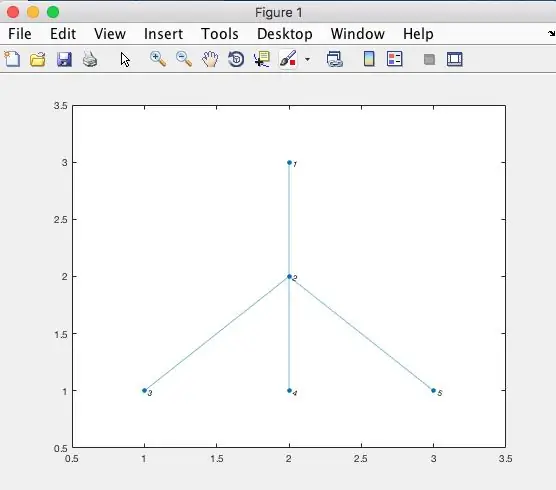
a = [0, 1, 0, 0, 0; 1, 0, 1, 1, 1; 0, 1, 0, 0, 0; 0, 1, 0, 0, 0; 0, 1, 0, 0, 0] %plottning g = diagram (a); %med hjälp av graffunktionen (grafteori) figur (1); h = plot (g);
Se figur 1 (ovan) för att se hur du lägger till kanter i angränsningsmatrisen med hjälp av koden i "Obs".
Steg 2: Skapa relationer
Nu när personerna (hörn eller noder) skapas måste vi skapa ett nätverk av relationer (linjer eller kanter på grafen). Detta kommer att simulera hur människor interagerar och träffar andra människor under en dag.
Detta kan göras på många sätt. Ett sätt att slutföra denna uppgift är att först tilldela varje person ett slumpmässigt nummer för att avgöra hur många människor varje person kommer att interagera med på en dag.
numOfFriendsMatrix = randi ([leastFriendsPersonCanHave, mostFriendsPersonCanHave], 1, NUMOFPEOPLE);
Detta gör en 1 till 20 matris av slumpmässiga heltal som representerar antalet interaktioner varje person har en dag. Kolumnerna i denna matris skulle vara det tal som motsvarar varje person. Om vi till exempel tilldelar minstFriendsPersonCanHave = 2 och mostFriendsPersonCanHave = 5 skulle vi få slumpmässiga värden mellan 2 och 5.
Har du problem med randi ()? Skriv i terminalen
hjälp randi
Därefter gör vi en randomiserad matris (kallad "allFriendsmatrix") av hur varje person i befolkningen är ansluten/interagerar inom befolkningen.
tempMatrix = ;
count = 0; allFriendsMatrix = ; för k = 1: NUMOFPEOPLE medan längd (tempMatrix) ~ = numOfFriendsMatrix (k) count = count +1; temp = randi ([1, NUMOFPEOPLE]); tempMatrix (count) = temp; avsluta varje gång längd (tempMatrix) ~ = 9 tempMatrix = [tempMatrix, NaN]; avsluta allFriendsMatrix = [allFriendsMatrix; tempMatrix]; tempMatrix = ; count = 0; slutet
Fördjupad förklaring av koden:
Först skapar vi en tom tillfällig matris för att hålla varje persons vänner/interaktionslista. Vi initierar också räkningen, som bara håller reda på var den nya slumpmässiga anslutningen ska hållas i tempMatrix. For -looparna går 20 gånger så att detta händer för varje enskild person i befolkningen. Den första medan loop går tills varje persons tempMatrix är lika lång som det slumpmässigt tilldelade antalet interaktioner. I denna slinga genereras ett slumpmässigt tal som motsvarar personen i befolkningen och placeras i tempMatrix. Eftersom längden på var och en av tempMatrixes är olika, behövde vi skapa några NaN -värden så att vi kan sammanfoga alla dessa tempMaticies till en matris ('allFriendsMatrix'). Den andra medan loop löser detta problem genom att lägga till NaN: er i varje tempMatrix. While loop var inställd på att köras 9 gånger eftersom det är ett nummer större än 5, vilket var den övre gränsen för vänner som en person kan tilldelas. Värdet "9" är variabelt och kan/måste ändras när "mostFriendsPersonCanHave" är större än 9. De tre sista kodraderna (exklusive slutet) lägger till tempMatrix till nästa rad i "allFriendsMatrix". Sedan rensar det ut tempMatrix och räknar för nästa person.
Produktion
Så här ska utgången se ut för den första körningen genom for -slingan (före de tre sista raderna).
tempMatrix = 16 8 17 16 13 NaN NaN NaN NaN
allFriendsMatrix =
16 8 17 16 13 NaN NaN NaN NaN 8 8 2 7 11 NaN NaN NaN NaN 10 13 NaN NaN NaN NaN NaN NaN NaN 11 17 2 NaN NaN NaN NaN NaN NaN 10 12 NaN NaN NaN NaN NaN NaN NaN NaN 4 13 2 12 NaN NaN NaN NaN NaN 17 10 9 3 1 NaN NaN NaN NaN 16 16 6 NaN NaN NaN NaN NaN NaN 3 8 17 17 14 NaN NaN NaN NaN 20 19 3 NaN NaN NaN NaN NaN NaN 13 10 NaN NaN NaN NaN NaN NaN NaN 2 19 18 10 16 NaN NaN NaN NaN NaN 2 6 14 3 13 NaN NaN NaN NaN 8 16 14 8 NaN NaN NaN NaN NaN 7 7 NaN NaN NaN NaN NaN NaN NaN 19 10 9 NaN NaN NaN NaN NaN NaN 10 19 NaN NaN NaN NaN NaN NaN NaN 5 18 NaN NaN NaN NaN NaN NaN NaN 1 7 NaN NaN NaN NaN NaN NaN NaN 16 7 13 10 1 NaN NaN NaN NaN
Lägg sedan till dessa relationer till adjMatrix.
för eachRow = 1: NUMOFPEOPLE
för eachCol = 1: 9 if isnan (allFriendsMatrix (eachRow, eachCol)) == 0 adjMatrix (eachRow, allFriendsMatrix (eachRow, eachCol)) = 1; adjMatrix (allFriendsMatrix (eachRow, eachCol), eachRow) = 1; slutet slutet slutet
Kod Förklaring
Denna dubbla för loop går igenom varje rad och kolumn i 'allFriendsMatrix'. If -satsen körs för alla värden som inte är 'NaN'. I grund och botten kommer det att skapa kanterna eller linjerna i grafen. Så den första raden detta kommer att göra är person 1 till person 16 och person 16 till person 1. Eftersom det inte är riktat måste 1 ändras för båda! Vi kan inte bara ha kanten 1 till 16 och inte 16 till 1. De måste vara symmetriska för att den ska fungera korrekt i Matlab.
I vår simulering konstaterade vi att människor inte kan interagera med sig själva. När vi randomiserade värdena finns det en chans att vår intilliggande matris har detta fel.
Låt oss fixa detta med följande kod:
för varje = 1: NUMOFPEOPLE
adjMatrix (varje, varje) = 0; slutet
Kod Förklaring
Detta för loop säkerställer att person 1 inte är ansluten till person 1, person 2 är inte ansluten till person 2, etc genom att göra dem alla 0. Som du kan se nedan i utdatasektionen har vi kvadratens diagonal matrisen uppifrån till vänster till nedre högra är alla 0: or.
Produktion
Detta är den sista adjMatrix för den aktuella simuleringen. Detta står för alla rader i diagrammet (figur 2).
adjMatrix =
0 0 0 0 0 0 1 1 0 0 0 0 1 0 0 1 1 0 1 1 0 0 0 1 0 1 1 1 0 0 1 1 1 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 1 1 0 0 1 0 0 0 0 0 0 0 0 1 0 0 0 1 0 0 0 0 1 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 1 0 1 0 0 0 0 0 1 0 0 0 1 0 1 0 0 0 1 0 0 0 1 1 0 0 0 0 0 0 0 1 1 1 0 0 0 0 0 1 1 0 0 0 0 1 0 1 0 1 1 1 1 0 0 0 1 0 0 1 0 0 0 0 1 0 1 0 0 0 0 0 0 1 0 0 0 1 1 0 0 0 0 0 1 0 1 1 0 0 0 0 0 1 0 1 0 1 0 0 0 1 1 0 0 0 1 1 0 1 1 0 1 0 1 0 0 0 0 0 1 0 0 1 0 0 0 0 0 0 0 0 1 0 0 1 1 0 0 0 1 0 0 0 0 0 1 0 1 0 0 1 1 1 0 0 1 0 0 0 0 1 0 0 1 0 0 0 0 0 1 0 0 0 0 0 0 0 1 1 0 0 0 1 0 0 1 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 1 1 1 0 1 0 1 0 0 0 0 1 1 1 0 0 1 0 0 1 0 1 1 0 0 0 0 0 0 0 0 1 0 0 0 0 0 1 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 1 0 0 0 0 0 1 0 0 1 0 0 0 0 0 1 1 0 0 0 1 0 0 0 0 0 1 0 0 1 0 0 1 0 0 1 0 0 0 0
Se figur 2 för att se grafen över 'adjMatrix'.
Steg 3: Lägg till sjukdomsstatistik
Nu när ditt program kan skapa en graf med en uppsättning slumpmässiga människor och skapa slumpmässiga relationer, måste vi ange sjukdomens information eller statistik för att se hur dessa interaktioner inom en befolkning kan öka eller minska infektionen.
Skapa dessa variabler:
unvacc %typ: dubbel; procents chans att ovaccinerade inte får sjukdomen
vaccintyp: dubbel; procents chans att vaccinerade inte får sjukdomen unvacc_perc %typ: dubbel; procent befolkning ovaccinerad init_infect %type: int; procent befolkningen vaccinerad
Därefter måste vi göra några beräkningar.
Vi ska göra en 'infectMat' som är en 3*NUMOFPEOPLE -matris.
vacc_perc = 1-unvacc_perc;
infectMat = nan (3, NUMOFPEOPLE); nummer = rund (vacc_perc * NUMOFPEOPLE); infektionsmat (1, 1: antal) = vacc; infektionMat (1, nummer+1: slut) = unvacc; infectMat (2, 1: end) = 0; infectMat (2, 1: init_infect) = 1;
Kod Förklaring
rad 1: Procent av befolkningen ovaccinerad beräknad
rad 2: skapa en 3*N antal personer matris
rad 3: ta reda på antalet personer som vaccinerats från vaccinerad andel
rad 4: ge dem en immunitet i samband med vaccinationen för de vaccinerade. Detta värde tilldelas baserat på forskning om sjukdomen.
rad 5: för resten av befolkningen (ovaccinerade personer), ge dem procentuell immunitet. Detta värde tilldelas baserat på forskning om sjukdomen.
rad 6: ställ inledningsvis alla människor till att inte smittas.
rad 7: för antalet personer som initialt smittats, fyll i de första par kolumnerna i enlighet därmed.
Nu när vi har ställt in alla parametrar för sjukdomssimuleringen kommer vi att slumpmässigt chansera om personen (både vaccinerad och ovaccinerad) blir smittad. Detta görs i nästa steg genom att tilldela slumpmässiga värden mellan 0 och 1 till varje person i den tredje raden i denna 'infectMat'.
Steg 4: Slumpmässigt chansen att en vaccinerad och ovaccinerad person kan bli smittad
Tilldela sedan varje person ett slumpmässigt nummer, detta kommer att användas senare för att avgöra om personen blir smittad eller inte.
för w = 1: längd (infectMat)
infectMat (3, w) = rand; slutet
Kodförklaring
Detta för loop handlar om den tredje raden av 'infectMat' som skapades i det sista steget. 'rand' tilldelar ett värde mellan 0 och 1 till varje index i rad 3.
Produktion
infektionMat är nu klar! Detta var med en befolkning med 100% vaccination och 1 person initialt infekterad.
infektionsmat =
Kolumnerna 1 till 12 0,7500 0,7500 0,7500 0,7500 0,7500 0,7500 0,7500 0,7500 0,7500 0,7500 0,7500 0,7500 1,0000 0 0 0 0 0 0 0 0 0 0 0 0,0869 0,5489 0,3177 0,9927 0,7236 0,5721 0,7172 0,9766 0,4270 0,9130 0,8973 0,8352 Kolumner 13 till 0,0075 0,7500 0,7500 0 0 0 0 0 0 0 0 0,0480 0,3593 0,2958 0,6291 0,1336 0,3740 0,8648 0,2503
rad 1: Procentuell chans att INTE få sjukdomen
rad 2: Infekterad eller inte infekterad (booleskt värde)
rad 3: Nummer som används för att kontrollera om en person som inte är smittad blir smittad om de träffar en infekterad person. Om oinfekterad person träffar en infekterad person är detta antal större än antalet i rad 1 (för samma kolumn), då är de infekterade. Vi kommer att koda denna funktionalitet i steg 7.
Steg 5: Skapa matriser för människor som är ovaccinerade och infekterade från initial information
Skapa två matriser som kallas "matrixUnvacc" och "matrixInfected" som lagrar alla infekterade personer från infectMat. Detta kommer att användas så att vi kan färgkoda grafen för dem som är infekterade, ovaccinerade eller vaccinerade, vilket hjälper till att visualisera effekterna av ovaccinerade kontra vaccinerade individer.
rensa varje
matrixInfected = ; matrixUnvacc = ; för h = 1: längd (infektionsmat) om infektionsmat (1, h) == unvacc matrixUnvacc = [matrixUnvacc, h]; ändänd för person = 1: NUMOFPEOPLE om infectMat (2, person) == 1 matrixInfected = [matrixInfected, person]; slutet slut
Kod Förklaring
Skapa två tomma matriser för att lagra antalet personer som är ovaccinerade respektive infekterade. Både för loopar körs 20 gånger och om if -satsen uppfylls, läggs numret till rätt matris.
Produktion
matrixUnvacc =
matrixInfected =
[1]
Steg 6: Plotta inledande diagram
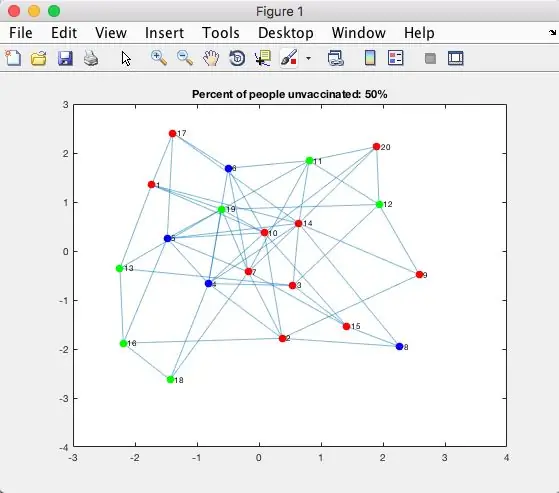
Därefter ska vi plotta adjacensmatrisen.
g = graf (adjMatrix);
figur (1) p = plot (g, 'NodeColor', 'b', 'MarkerSize', 7); highlight (p, matrixUnvacc, 'NodeColor', 'g') highlight (p, matrixInfected, 'NodeColor', 'r') title_unvacc = unvacc_perc*100; title (['Procent av personer som inte är vaccinerade:', num2str (title_unvacc), '%']); paus (hastighet)
Kodförklaring
Grafteori i Matlab har inbyggda funktioner. När vi använder grafen () -funktionen kan vi översätta 'adjMatrix' till en verklig oriktad graf. Vi måste sedan skapa en plot med funktionen plot () för att faktiskt se hur det ser ut. Vi ställer in denna tomt () till en variabel så att vi lättare kan manipulera och ändra färgerna på tomten under hela simuleringen. Alla människor (eller noder) är initialt inställda på färgen "blå". Därefter är alla ovaccinerade människor inställda på färgen "grön". De infekterade personerna ställs sedan in i färgen "röd". Titeln är inställd enligt ett visst procentuellt värde för ovaccinerade personer som testas. Pausfunktionen () stoppar tillfälligt MatLab -körningen. Vi passerar genom den variabla hastigheten som sprids som beräknas i sekunder.
Se bild (ovan) för att se en slumpmässig färgkodad graf.
Läs mer om funktionen highlight () i MatLab.
Steg 7: Simulera utvecklingen av infektionen
Därefter måste vi ta reda på vem som smittas efter interaktionerna (inspelade i adjMatrix) och uppdatera grafen när någon blir infekterad.
Använd adjMatrix för att avgöra vilka personer som är smittade efter deras interaktioner med människor på en dag.
för eachRow = 1: längd (adjMatrix)
if infectMat (2, eachRow) == 1 för eachCol = 1: length (adjMatrix) if adjMatrix (eachRow, eachCol) == 1 % eachRow = personen % eachCol = dess vän % varje persons vän och se om de är infekterade. if infektionMat (3, eachCol)> infektionMat (1, eachCol) infektionMat (2, eachCol) = 1; markera (p, eachCol, 'NodeColor', 'r') paus (hastighet) slutet slutet slutet slutet slutet
For loop slingorna går igenom varje person. Den kontrollerar att om personen är infekterad kommer den att kontrollera var och en av de personer/vänner de interagerade med och kontrollera om vänens immunitetsnivå var större än sjukdomens styrka. Det är här 'infectMat' vi skapade tidigare spelar in. Den första och tredje raden i varje kolumn i vännen jämförs och om den tredje raden är större betyder det att vännen inte hade tillräckligt hög immunitet för att undkomma sjukdomen och i slutändan blir smittad. Vi byter också till färg med markering () till rött om de blir smittade.
Nu ska din kod för simuleringen fungera! och för vilken befolkningsstorlek som helst, ändra bara NUMOFPEOPLE!
Steg 8: Använd Monte Carlo -teorin
För att ta detta ett steg längre och extrahera data från vår simulator ('infektionSim.m') ville vi beräkna och diagramma trenden i procent av ovaccinerade som smittades och andelen vaccinerade som smittades. Vi antar att andelen vaccinerade som smittats bör vara mycket lägre än andelen icke -vaccinerade som smittats.
Steg 9: Gör filen ('infectSim.m') med simuleringen i en funktion
För att köra Monte Carlo skulle vi vilja köra simuleringen flera gånger och samla in data så att vi kan använda det för att kartlägga andelen personer som smittades.
Funktionen kan ställas in så här:
function output = infectSim (unvacc, vacc, NUMOFPEOPLE, unvacc_perc, init_infect, speed)
Kommentera variablerna i din simulering eftersom du nu skickar in dessa genom huvudfilen (vi börjar skriva detta i steg 12):
unvacc, vacc, NUMOFPEOPLE, unvacc_perc, init_infect
Den nya variabeln
fart
kommer att tilldelas i huvudfilen (Monte_Carlo.m).
Obs: Glöm inte slutet längst ner i funktionsfilen för att avsluta funktionen!
Steg 10: Beräkna andelen ovaccinerade och vaccinerade som smittats
Detta beräknar andelen ovaccinerade som smittats. Den här koden går längst ner i filen 'infectSim.m'.
number_of_unvacc = 0;
number_of_infec_unvacc = 0; %beräknar andelen ovaccinerade personer som smittades för x = 1: length (infektionsmat) om infektionsmat (1, x) == unvacc nummer_of_unvacc = nummer_of_unvacc+1; avsluta om infektionsmat (1, x) == unvacc & infektionsmat (2, x) == 1 nummer_of_infec_unvacc = nummer_of_infec_unvacc +1; slutet slutet percent_of_unvacc_and_infec = (number_of_infec_unvacc / number_of_unvacc)*100;
Kodförklaring
I for -loop kommer den att loopa över NUMOFPEOPLE gånger. Varje gång antalet i infektionMat motsvarar unvacc -numret (dvs. 0,95 == 0,95), kommer antalet ovaccinerade personer att ökas med 1. Varje gång antalet i infektionsmatten motsvarar unvacc -numret och de är infekterade, kommer antalet infekterade och ovaccinerade ökar med 1. Den sista raden delar antalet infekterade, ovaccinerade personer med det totala antalet ovaccinerade. Sedan beräknas procentsatsen utifrån detta.
Utmaning:
Försök att beräkna andelen vaccinerade personer som smittats! (Tips: den liknar den här koden ovan, men vissa variabler ändras och namn justeras.)
Därefter beräknas andelen infekterade utifrån den totala befolkningen:
pre_per_infect = cumsum (infectMat (2,:));
per_infect = (pre_per_infect (1, NUMOFPEOPLE)/NUMOFPEOPLE)*100;
Kodförklaring
Den kumulativa summan beräknas med hjälp av den andra raden i infectMat, som lagrar 1: or och 0: or beroende på om personen är infekterad eller inte. Eftersom cumsum () -funktionen ger tillbaka en matris, tar vi det sista värdet i matrisen ('pre_per_infect (1, NUMOFPEOPLE)'), vilket ska vara den faktiska summan av alla värden från 'infectMat (2,:)'. Genom att dela summan med NUMOFPEOPLE och multiplicera den med 100 får vi den sista andelen smittade i den totala befolkningen.
Steg 11: Skapa en utmatningsvariabel i din funktion 'infectSim.m'
output = [per_infect, percentage_of_unvacc_and_infec, percentage_of_vacc_and_infec];
Kodförklaring
Spara denna information i utdata, som skickas tillbaka till main (Monte_Carlo.m) när funktionen anropas och körs. Dessa data används för att räkna ut poängen i procent av de infekterade av dem som är vaccinerade och ovaccinerade.
Din "infectSim.m" -funktion bör göras nu! Det kommer dock inte att köras eftersom vi fortfarande behöver skriva huvudet!
Steg 12: Skapa en meny för att få de första villkoren för simuleringen från användaren
Kom ihåg hur vi sa variabeln
fart
skulle skapas och passeras genom huvudfunktionen? Vi måste få värdena för att kunna övergå till funktionen. Observera, ordningen på värdena när du kallar funktionen spelar ingen roll!
Börja med att be användaren att skriva några svar i terminalen.
> Välj en sjukdom. Observera att det är skiftlägeskänsligt >> Kikhosta >> Influensa >> Mässling >> Sjukdom vald: Influensa >> Välj befolkningsstorlek. >> 20 >> 200 >> Vald befolkning: 20 >> Välj simuleringshastighet. >> Snabb >> Långsam >> Vald hastighet: Snabb
Denna kod nedan frågar användaren vilken sjukdom de vill undersöka.
disp ('Välj en sjukdom. Observera att den är skiftlägeskänslig')
fprintf ('Pertussis / nFlu / nMeasles / n') disease = input ('Disease Chosen:', 's'); om isequal (sjukdom, 'Kikhosta') vacc =.85; %15 procent chans att få sjukdom unvacc =.20; %80 procent chans att få sjukdom annars om det är ojämnt (sjukdom, 'influensa') vacc =.75; %25 procent chans att få sjukdom unvacc =.31; %69 procent chans att få sjukdom annars om det är ojämnt (sjukdom, "mässling") vacc = 0,97; %3 procent chans att få sjukdom unvacc =.10; %90 procent chans att få sjukdomen slut
Kodförklaring:
Disp () -funktionen skriver ut uttalandet till skärmen och det skriver också ut de olika alternativen. Sjukdomen kommer att tilldelas i enlighet därmed. Denna version tar för närvarande inte hänsyn till ogiltig inmatning. Ogiltig inmatning ger ett fel och stoppar programmet helt. Varje sjukdom har vacc- och unvacc -värden associerade med den. Dessa värden är INTE slumpmässiga. Vi fick dessa värden från att undersöka statistik om sjukdomarna.
Därefter måste vi fråga användaren om de vill testa en stor eller liten befolkningsstorlek för sin valda sjukdom.
disp ('Välj befolkningsstorlek.')
fprintf ('20 / n200 / n ') speed = input (' Befolkning vald: ',' s '); if isequal (speed, '20') population_size = 20; elseif isequal (hastighet, '200') population_size = 200; slutet
Kodförklaring
Detta skriver ut ett uttalande till användaren och uppmanar användaren att ange vilken befolkningsstorlek den vill testa. Denna version tar för närvarande inte hänsyn till ogiltig inmatning. Ogiltig inmatning ger ett fel och stoppar programmet helt. 20 valdes eftersom det är en liten urvalsstorlek som fortfarande ger en god uppfattning om hur infektion sprids över en liten befolkning. 200 personer valdes som det större alternativet eftersom 200 punkter på diagrammet knappt hade någon överlappning av punkter så att allt lätt kunde ses och särskiljas från varandra.
Därefter måste vi hitta simuleringens hastighet.
disp ('Välj simuleringshastighet.')
fprintf ('Fast / nSlow / n') speed = input ('Speed hastighet:', 's'); om isequal (hastighet, 'Snabb') sim_speed = 0; elseif isequal (hastighet, 'Slow') sim_speed = 0,25; slutet
Kodförklaring
Denna process var densamma som att få typen av sjukdom och befolkningens storlek. För snabbt blir det ingen paus. och för långsam blir det en 0,25 sekunders fördröjning i for -slingan när simuleringen körs.
Bra! Nu har vi alla inmatningar från användaren vi behöver! Låt oss gå vidare till att samla in data för olika procentsatser av ovaccinerade människor.
Steg 13: Välj en procentandel av ovaccinerade personer och beräkna genomsnittet av ovaccinerade och infekterade för vald procentandel
Denna kod är för 0% av de ovaccinerade.
% ------- % 0 Ovaccinerad ------------
per_infect_av_0 = ; percentage_of_unvacc_and_infec_av_0 = ; för i = 1:20 ut = infektionSim (unvacc, vacc, population_size, 0, 1, sim_speed); per_infect_av_0 = [per_infect_av_0, out (1, 1)]; percentage_of_unvacc_and_infec_av_0 = [percent_of_unvacc_and_infec_av_0, out (1, 2)]; slut genomsnittlig_infekterad_0 = medelvärde (per_infekt_av_0); average_unvacc_and_infected_0 = medelvärde (percentage_of_unvacc_and_infec_av_0);
Kodförklaring:
For -slingan körs 20 gånger. Utmatningen från funktionen, infectSim (), lagras in out. Varje gång for loop -löpningen körs, läggs andelen infekterade i den totala populationen till matrisen 'per_infect_av_0'. Dessutom läggs även andelen ovaccinerade och infekterade till varje gång i matrisen 'percentage_of_unvacc_and_infec_av_0'. På de två sista raderna genomsnitts dessa två ovan nämnda matriser och lagras i variabler. Sammanfattningsvis lagras procentsatserna för varje simulering, i genomsnitt och diagram. Monte Carlo används för att visa medelvärdet för att köra en simulering och visa resultatet. För våra experimentella ändamål väljer vi att köra simuleringen 20 gånger och genomsnittliga dessa värden.
Utmaning:
Upprepa för alla procentsatser du vill testa! Detta kan göras genom att ändra variabelnamnen enligt procenttalen. Vi testade för 0%, 5%, 10%, 20%, 30%och 50%.
Antydan:
Den enda raden som måste ändras i den faktiska koden är
out = infectSim (unvacc, vacc, population_size, 0, 1, sim_speed);
Ändra nollan till procenten i decimalform. Till exempel, för 5% ovaccinerad simulering, bör 0: n ersättas med 0,5.
Steg 14: Graf: 'Trenden för infektion i ovaccinerade vs. Vaccinerad för specificerad sjukdom '
Detta är koden för att göra en graf över utvecklingen av infektion hos icke -vaccinerade personer mot icke -vaccinerade personer.
graph_mat_y = [average_infected_0, average_infected_5, average_infected_10, average_infected_20, average_infected_30, average_infected_50];
graph_mat_x = [0, 5, 10, 20, 30, 50]; lutning = (genomsnittlig_infekterad_5-genomsnittlig_infekterad_0)/5; line_y = [genomsnitt_infekterad_0, (lutning*50)+genomsnittlig_infekterad_0]; line_x = [0, 50]; figur (2) plot (graph_mat_x, graph_mat_y); line (line_x, line_y, 'Color', 'red', 'LineStyle', '-'); title (['Trend i ovaccination för', sjukdom]); xlabel ('Procentandel initial ovaccinerad'); ylabel ('Procentandel av sista infekterade')
Kodförklaring
rad 1: tilldelade y -värden till medelvärdena för procent infekterade
rad 2: tilldelade x -värden till andelen initiala procent som inte är vaccinerade
rad 3: beräkna lutningen på 0% och 5%
rad 4: lagra y -värden för rad. Detta är en fortsättning på avsnittet 0% till 5%.
rad 5: lagra y -värden för rad. Denna rad sträcker sig över grafens längd.
rad 6: skapa figur
rad 7: rita upp grafen x och y -värdena för de procent smittade, som är ovaccinerade.
rad 8: rita linjen. Detta används för att visa att det inte ökar linjärt, utan exponentiellt.
rad 9: Ange titel för diagrammet.
rad 10-11: Ange x- och y-etiketter för diagram.
Nu borde du kunna se att ju större andel av befolkningen som inte är vaccinerad, desto större infektionsmängder. Du kommer också att se att de flesta prickarna som blir röda är gröna prickar, vilket visar att vaccinet hjälper till viss del! Hoppas du gillade denna handledning. Kommentera om du har några frågor!
Steg 15: Slutprodukt: Hur simuleringen ser ut
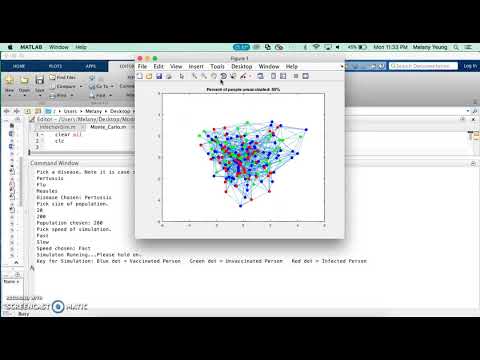
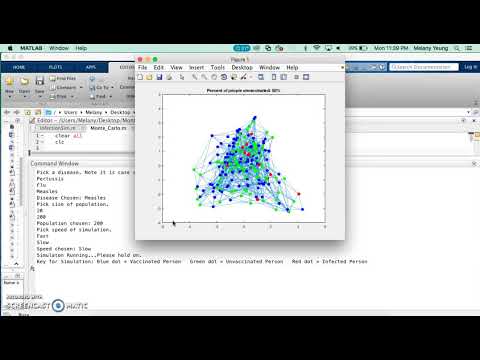
All kod finns här
Rekommenderad:
Få blinda att känna igen saker genom att röra saker runt dem med MakeyMakey: 3 steg

Gör blinda igenkänna saker genom att röra saker runt dem genom att använda MakeyMakey: introduktion Detta projekt syftar till att göra blindas liv enkelt genom att identifiera saker runt dem genom beröringssinne. Jag och min son Mustafa funderade på att hitta ett verktyg för att hjälpa dem och under den period som vi använder MakeyMakeys hårdvara
DIY MusiLED, musiksynkroniserade lysdioder med ett klick Windows och Linux-applikation (32-bitars och 64-bitars). Lätt att återskapa, lätt att använda, lätt att porta .: 3 steg

DIY MusiLED, musiksynkroniserade lysdioder med ett klick Windows och Linux-applikation (32-bitars och 64-bitars). Lätt att återskapa, lätt att använda, lätt att porta .: Detta projekt hjälper dig att ansluta 18 lysdioder (6 röda + 6 blå + 6 gula) till ditt Arduino-kort och analysera datorns ljudkortets realtidsignaler och vidarebefordra dem till lysdioderna för att tända dem enligt slageffekterna (Snare, High Hat, Kick)
Hacka Mac -bärbar dator för att bli en Mac -surfplatta på 15 minuter eller DIY Cintiq: 6 steg (med bilder)

Hacka Mac -bärbar dator för att vara en Mac -surfplatta på 15 minuter eller DIY Cintiq: Jag har fått många frågor från platser där detta har dykt upp: 1. ja det är tryckkänsligt2. det fungerar bara där min wacom -sensor är … i princip det vita blocket i videon. Jag använde g4 eftersom det har ett trasigt moderkort och var virtuellt
Programmera din dator att stängas av automatiskt genom att ange vilken tid du vill att den ska göra det: 4 steg
Programmera din dator för att stänga av automatiskt genom att ange vilken tid du vill att den ska göra det: hej, det är en annan instruktion som tagits från mitt dagliga liv … förra gången var jag tvungen att ladda ner massor av programvara till min dator och jag var tvungen att låta den ladda ner över natten ville jag inte ha datorn påslagen hela natten efter att jag hade laddat ner och vid
Ett riktigt enkelt/enkelt/inte komplicerat sätt att få människor/människor/djur/robotar att se ut som om de har riktigt cool/ljus värmevision (valfri färg) med GIMP: 4 steg

Ett riktigt enkelt/enkelt/inte komplicerat sätt att få människor/människor/djur/robotar att se ut som om de har riktigt cool/ljus värmevision (valfri färg) med GIMP: Läs … titeln
