Innehållsförteckning:
- Författare John Day day@howwhatproduce.com.
- Public 2024-01-30 12:45.
- Senast ändrad 2025-01-23 15:11.
Av: Madhumita Kannan, Henry Nguyen, Ashley Urrutia Avila, Mei Jin
Denna MATLAB -kod är ett program för att upptäcka den exakta storleken, formen och platsen för en tumör som finns i en patients hjärnans MR -skanningar. Detta program är utformat för att ursprungligen fungera med tumörupptäckt i hjärnans MR -skanningar, men det kan också användas för cancerdiagnostik i andra organsökningar.
Följande instruktioner beskriver först metoderna för bildanalys genom filtrering och rengöring av MR -skanning, genom binärisering, medianfiltrering och skjutfönster. Därefter kommer den att instruera om hur man isolerar tumören med en förgenererad elliptisk mask och filtrerar den vidare för att beskriva omkretsen av tumörens form.
När tumören upptäcks kommer instruktionerna ytterligare att beskriva hur detta program ska införlivas i ett grafiskt användargränssnitt (GUI). Genom dessa instruktioner bifogas lämplig kod och filer för att förklara hur denna MR -skanningsanalys fungerar.
Några saker att veta, ladda ner och ha klart innan du fortsätter med denna instruerbara: 1. Se till att ha den senaste versionen av MATLAB nedladdat. Du kan installera R2018b här:
2. För att kunna köra detta program måste du ha åtkomst till MR -hjärnskanningsfiler. Även om vissa alltid kan hittas från Googles bilder, kan noggrann och noggrann analys utföras från korrekta bilder av olika lager av hjärnskanningar för varje patient. Du kan komma åt filerna för 20 olika patienter med glioblastom före och efter behandling från denna databas:
3. Fokus för detta program och de olika metoderna som styr detta projekt beskrivs i detta forskningsarbete:
Steg 1: Starta grafiskt användargränssnitt (GUI)
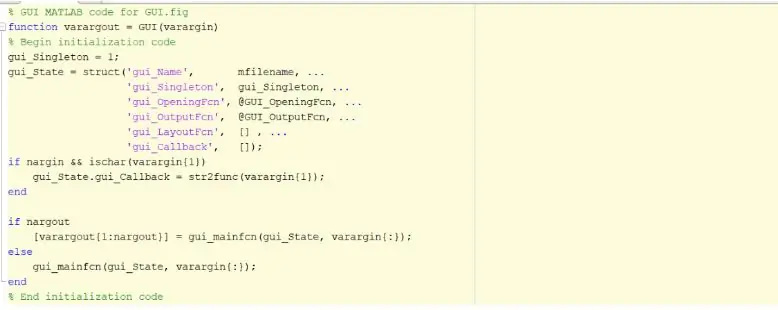
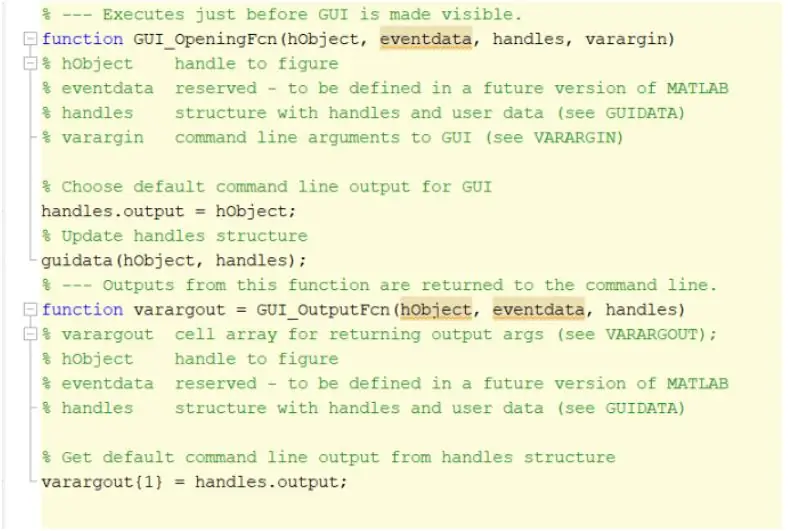
Det första steget skulle vara att skapa och initiera det grafiska användargränssnittet, GUI. Detta kan göras genom att skriva in guiden i kommandofönstret, trycka på enter och skapa ett nytt GUI. När detta steg är slutfört kan du börja skapa funktioner som axlar, statisk text, redigera text och tryckknappar som visas när programmet körs och användaren kan interagera med. Dessa funktioner kan redigeras och manipuleras via fastighetsinspektören, men den viktigaste funktionen som måste ändras när du skapar dessa funktioner är taggnamnet. Det är viktigt att ändra taggnamnet för varje funktion som implementeras eftersom det gör att vi kan skapa en urskiljbar återuppringningsfunktion. När du är nöjd med layouten på ditt GUI kan du fortsätta att ladda DICOM -filerna som kommer att visas i GUI.
Steg 2: Ladda och läsa MR -bilder i MATLAB
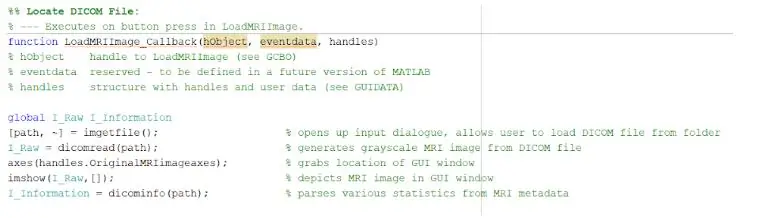
För att ladda DICOM -filer måste du korrekt initiera återuppringningsfunktionen som skulle utföras när du trycker på knappen "Ladda MRI -bild." När detta är klart måste du skapa en global variabel som visar bilden på handtagen axlar där du vill att den ursprungliga MRI -bilden ska visas. MRI -skanningsbilderna som laddas ner från databasen är alla DICOM -formaterade filer som måste laddas in i din MATLAB -katalog. Leta reda på filen med imgetfile för att ladda dem i programmet. Bilderna läses med den inbyggda MATLAB -funktionen 'dicomread', och den första råa bilden för varje fil är inbäddad i de vänstra GUI -axlarna med hjälp av imshow.
Den inbyggda MATLAB -funktionen 'dicominfo' är också extremt användbar för att hantera all information för varje MRI -dicom -fil. Vi använde denna funktion för att extrahera all beskrivande information om patienterna, till exempel deras kön, ålder, vikt och längd. Denna funktion ger dig också stapelordningen som är användbar för implementering av programmet i det grafiska användargränssnittet. Vi skapade variabler för var och en av den beskrivande informationen för patienterna som kommer att användas för GUI när detekteringsknappen trycks in.
Steg 3: Bildfiltrering

När DICOM -filen för den råa bilden har laddats och lästs måste bilden konverteras från gråskala till en binär form som består av endast svartvita pixlar Vi använde funktionen 'imbinarize' för att skapa en binär bild från den råa bilden med kontrollera aspekter av adaptiv tröskelvärde vid känslighetsvärde på 0,59. Standardtröskelkänslighetsfaktorn 0,5 var låg och kunde inte upptäcka de ljusare fläckarna och fläckarna från bilden, så vi ökade den till 0,59.
Den binäriserade bilden bearbetas sedan genom ett medianfilter med funktionen 'medfilt2' eftersom den binära bilden är tvådimensionell. Vi ställer in varje utgångspixel för att innehålla medianvärdet i 5 x 5 -grannskapet runt motsvarande pixel i den inmatade binära bilden. Detta minskar bruset och bevarar kanterna i en 5 x 5 kvadrat runt varje pixel. Därefter applicerar vi ett skjutfönster med "strel" för att skapa ett diskformat platt strukturelement med en stadsradie på 2 för att identifiera varje central, ursprungspixel, i varje diskområde. Vi använde ett diskstruktureringselement eftersom vi analyserar varje cirkulär plats och pixlarna inom varje plats, så ett diskformelement är mer användbart.
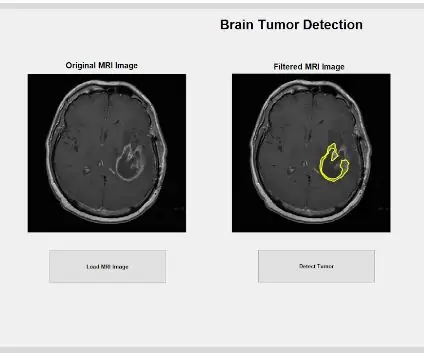
När bilden har filtrerats kan den rengöras med funktionen "stäng" för att ta bort de svarta fläckarna mellan de filtrerade vita pixlarna i bilden och stänger alla luckor runt den. Den fullständigt bearbetade bilden kan sedan plottas i den andra delplanen i den förtilldelade figuren, vilket möjliggör en jämförelse mellan den råa och filtrerade bilden.
Steg 4: Tumörisolering genom elliptisk mask


Tumörens ljuspunkter kan sedan isoleras från den huvudsakliga filtrerade bilden genom en förgenererad elliptisk mask. För att skapa den här masken bör du känna till storleken på den ursprungliga, råa MR-skanningsbilden, och med hjälp av dess rad- och kolumnlängd, som x respektive y- koordinater, allokera mittkoordinaterna för elliptiska. Vi ställer in y-axeln som en huvudaxel med en radie på 50 enheter från mitten och den mindre axeln med en radie på 40 enheter från mitten.
Vi använde MATLAB-funktionen 'meshgrid' för att generera ett kartesiskt plan med tvådimensionella rutkoordinater baserat på koordinaterna i vektorerna från 1 till x-axelns längd och från 1 till längden på y-axeln i bilden. Col är en matris där varje rad är en kopia av x-axeln, och rad är en matris där varje kolumn är en kopia av y-axeln. Det kartesiska rutnätet som representeras av koordinaterna Col och Row har längd (1: Y_Size) rader och längd (1: X_Size) kolumner. Använd indexen för Col och Row som genereras av det kartesiska rutnätet för att bestämma ellipsekvationen beroende på den förutbestämda radien och mittkoordinaterna. Den elliptiska konturen kan nu fyllas med de vita pixlarna från tumörfläckar.
Med hjälp av den förgenererade elliptiska masken kan vi beskära den specifika tumören du vill analysera från den filtrerade bilden. Den elliptiska masken upptäcker vilka fläckar som logiskt passar in i ellipsens kontur och accepterar detta som en fläck på den filtrerade bilden för att vara acceptabel som en tumör. Funktionen 'bwareafilt' filtrerar sedan bort alla andra objekt utanför denna detekterade tumör från bilden. Vi använde ett specifikt fönster på 500 x 4000 empiriskt baserat på måtten på alla bilder. Vi applicerade sedan ett annat skjutfönster med "strel" som ett platt diskformat strukturelement med en större stadsradie på 6, för att stänga luckorna mellan varje central vit pixel i den detekterade tumören. Den upptäckta tumörpunkten städas ytterligare med hjälp av "stäng" för att ytterligare eliminera de svarta pixlarna och fylla i alla hål med "fyllning". Denna bearbetade tumör kan sedan visas i den tredje delplanen i den förfördelade tomten för att ge en jämförelse mellan den isolerade tumören och de ursprungliga och filtrerade bilderna av MR -skanningen.
Steg 5: Tumöröversikt
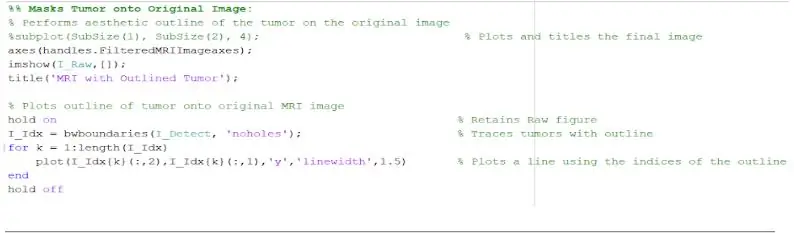
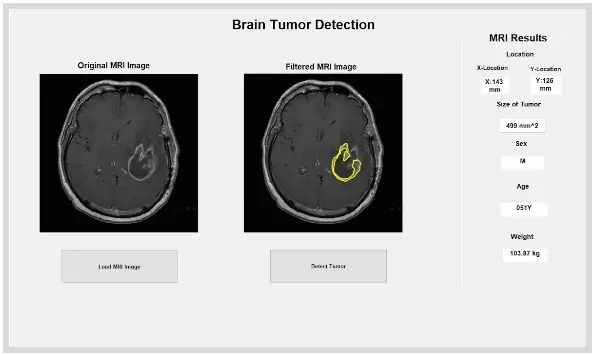
Nu när tumören isoleras med masken kan den skisseras och visas på originalbilden för att visa dess exakta plats. För att göra detta använde vi funktionen "bwboundaries" för att spåra den tidigare upptäckta tumören med en kontur. Vi specificerade konturen för att inte inkludera hålen i tumörobjektet som det skisseras. Detta kan ritas in på den ursprungliga, råa bilden, med hjälp av en "för" -slinga som plottar konturen runt tumören med hjälp av linjens index med en linjebredd på 1,5 pixlar. Denna kontur plottas sedan på den råa bilden, som visar tumörens exakta storlek och plats, i förhållande till den ursprungliga MR -skanningen.
Steg 6: Analysera de fysiska egenskaperna hos tumörer
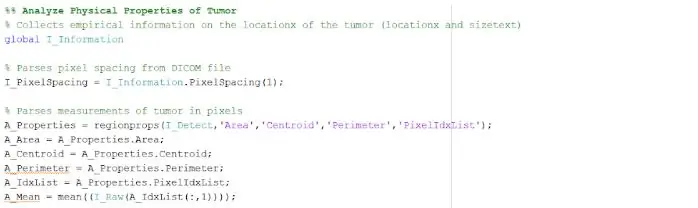
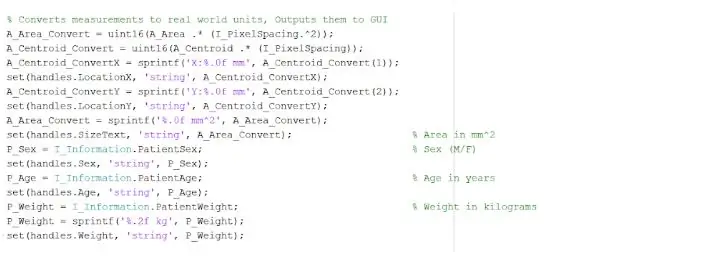
Den isolerade och skisserade platsen kan ge oss användbar information om tumörens storlek, yta och plats. Vi använde funktionen ‘regionprops’ för att upptäcka tumörens egenskaper som avser area, omkrets, centroider och pixelindexvärdet. Detta pixelindexvärde ger oss verkliga världsenheter för varje pixel i varje bild, unikt för varje genomsökning. Dessa egenskaper kan sedan omvandlas till verkliga enheter på millimeter. Den empiriska informationen som programmet ger oss är unik för varje MR -skanning och är oerhört användbar för att bestämma storlek, plats och typ av tumör som användarna kan analysera och införliva i det grafiska användargränssnittet.
Rekommenderad:
Kontrollera Raspberry Pi med Matlab: 5 steg

Kontrollera Raspberry Pi med Matlab: Hej, denna handledning handlar om att styra din huvudlösa hallon pi med matlab. Du kan behöva installera matlab till den senaste versionen för stöd för nyare hallon pi -kort
Använda MATLAB App Designer med Arduino: 5 steg

Använda MATLAB App Designer Med Arduino: MATLAB App Designer är ett kraftfullt verktyg som låter dig designa grafiska användargränssnitt (GUI) med alla MATLAB -funktioner. I denna handledning ska vi göra ett GUI för att styra ljusstyrkan på en LED genom ett enkelt att följa stegen. N
Hur man gör ett vuxenhörningstest med MATLAB: 6 steg

Hur man gör ett hörselprov för vuxna med MATLAB: DISCLAIMER: Vårt test är INTE en medicinsk diagnostik och bör inte användas som sådant. För att mäta hörseln noggrant, kontakta en läkare. Med hjälp av material som vi redan hade gjorde vår grupp ett hörselprov. Vårt test är endast avsett för vuxna och tonåringar
Styr Arduino trådlöst med MATLAB: 11 steg

Styr Arduino trådlöst med MATLAB: Har sett få DIY -appar om hur man upprättar en kommunikation mellan MATLAB -applikation och Arduino som är ansluten till PC. Jag har dock inte stött på något som styr Arduino via MATLAB trådlöst med ENC28J60 -kompatibel Ethernet -sköld
Roomba med MATLAB: 4 steg (med bilder)

Roomba med MATLAB: Detta projekt använder sig av MATLAB och en iRobot Create2 programmerbar robot. Genom att testa vår kunskap om MATLAB kan vi programmera Create2 för att tolka bilder och upptäcka signaler. Robotens funktionalitet beror huvudsakligen på
