Innehållsförteckning:
- Författare John Day day@howwhatproduce.com.
- Public 2024-01-30 12:38.
- Senast ändrad 2025-01-23 15:10.
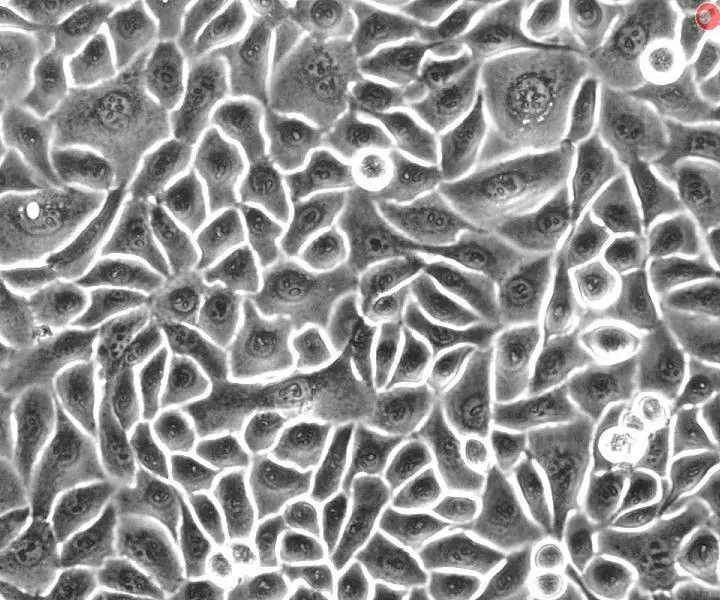
Vårt Sandbox -projekt syftar till att hjälpa forskare inom det biologiska området att analysera cellprover och ta reda på villkoren för deras celler. När användaren matat in en bild av sitt cellprov bearbetar vår kod bilden för att göra den redo för cellräkning genom att konvertera bilden till gråskala och binär. Koden använder tröskelvärde för att ta bort överflödig information som inte avser de faktiska cellerna för att exakt hitta cellmassorna. Efter att ha räknat antalet celler i bilden ger vår kod bildens pixelstorlek och hittar sedan cellens fördelning genom att rita antalet celler kontra cellens yta för att ge användaren sammanflödet, vilket är procent av ytan på en odlingsrätt som täcks av cellerna av intresse. Baserat på sammanflödet kan forskaren avgöra om cellerna ska passeras eller inte; passage av celler avser att utöka livslängden eller antalet celler eller mikroorganismer genom att överföra en del av kulturen till ett nytt tillväxtmedium, och det är användbart när celler blir för stora eller tar slut på mat. Efter att ha kört koden och bearbetat bilden kan användaren bestämma om de accepterar eller avvisar resultaten och fortsätter att justera tröskeln för bättre hämtning av data om det behövs.
Steg 1: Välj och konfigurera bild för analys
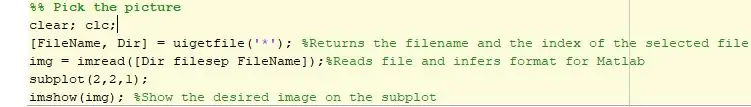
Det första steget i vår kod är att välja rätt bild och konfigurera den för analys i Matlab. Vi kan välja bilden med uigetfile -funktionen som gör att vi kan ta en fil och infoga den i programmet. Sedan läser vi bilden med imread och konfigurerar den för analys i Matlab. Bilden som väljs visas sedan på en delplan.
Steg 2: Tröskel och GUI
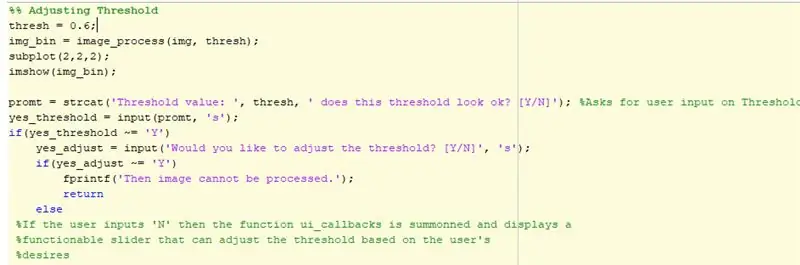
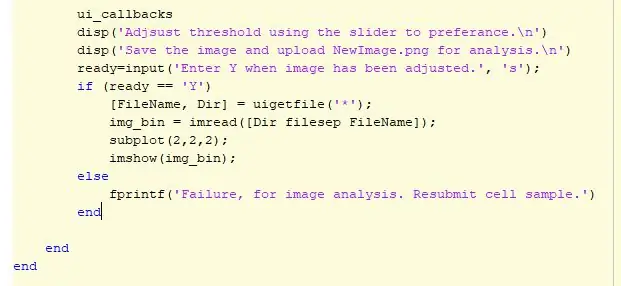
I början av koden väljs en bild med "uigetfile" och sedan definierar den bilden med en variabel. Variabeln kommer sedan att användas för att identifiera koden när olika analyser körs. En 2x2 delplan skapas i figur. I position 1 visas originalbilden. Nästa avsnitt i koden är där justeringar av trösklar sker. Initialt används en standard på ett tröskelvärde på 0,6 och visas i position 2 på delplanen. En if-sats används sedan för att avgöra om användaren vill behålla tröskeln eller justera den. Användaren kan än justera tröskeln med ett GUI som inkluderar bilden vid olika tröskelvillkor, reglaget och en spara -knapp. När tröskeln är inställd klickar användaren på knappen Spara för att spara bilden och den kommer att lagras i användarens MATLAB -filer som en-p.webp
Steg 3: Plotta konturer och celldistribution
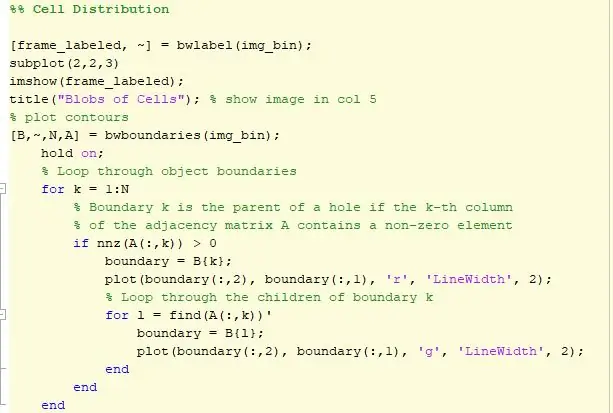
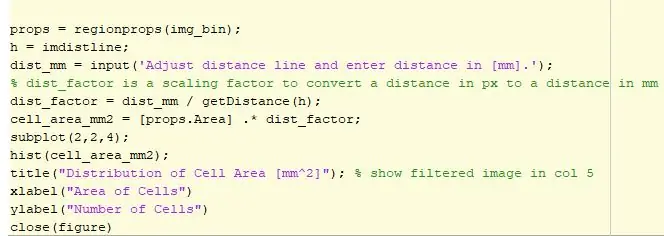
Nästa del av koden plottar konturerna. Det finns en slinga där cellerna är inneslutna av en röd omkrets, och de celler som finns på toppen av en annan cell är markerade med grönt. Den konturerade bilden visas sedan i position 3 med en interaktiv distanslinje. Den här raden bestämmer antalet pixlar i raden justerad av användaren för en pixel till millimeteromvandlare. Avståndsfaktorn multipliceras sedan med den yta som bestäms av regionprops och ytan uttrycks nu i millimeter i kvadrat. Data plottas sedan med hjälp av ett histogram för att se fördelningen av celler efter dess område. Detta histogram visas sedan i position 4.
Steg 4: Konvertera cellbild

I det här steget tog vi den gråskala bilden och binariserade, filtrerade och inverterade den. Genom att utföra dessa funktioner på bilden avlägsnades bullriga pixlar som kan misstas som celler och gjorde bilden smidigare och mjukare runt cellkanterna. Detta gjordes för att extrahera cellerna i bilden som urskiljbara "klumpar" som skiljde sig i intensitet från bakgrunden. "Blobs" var högintensiva vita bilder och bakgrunden var svart. Om det tillåts lite mer tid skulle vi ha använt en annan Blob -bildkonvertering istället för imbinarize -funktionen för att vara mer exakt och mer passande för våra bilder, men vi behövde mer tid för att forska och implementera funktionen.
Steg 5: Räkna celler och beräkna cellkonfluens

I det här steget i koden ville vi räkna antalet celler som fanns i bilden. Vi använde främst funktionen regionprops för att beräkna ytorna på blobbarna och om området beräknades vara i vår önskade gräns skulle det ritas på en delplot. Gränserna sattes för att ta bort små bullriga pixlar eller stora intensiteter som inte var celler. Cellräknaren skulle sedan räkna centroiderna som sedan ritades och lägga till dem i räknaren i for -slingan. När cellernas områden bestämdes kunde vi beräkna sammanflödet. Den viktigaste betydelsen av detta steg i koden var att hitta sammanflödet av cellerna och detta var avgörande för vårt slutmål med koden. Vi gjorde detta genom att beräkna genom att summera pixlarna (summa (allAreas)) i varje blob och sedan dela det med bildens totala pixelvärde (numel (img)). Detta förhållande skulle ge oss sammanflödet och om det bestämdes vara större än 80% än det är dags för cellerna att passeras av forskaren. Vi strävade efter att vara så exakta och exakta som möjligt men med den begränsade tiden så uppstod felaktigheter. Om tiden tillåter skulle vi ha tittat in på sätt att göra räkningen av klossar mer exakt, till exempel fler filtreringstekniker och/eller en Hough Transform eftersom det inte gjorts tillräckligt med forskning för att testa den filtreringstekniken
Steg 6: Cellernas rundhet
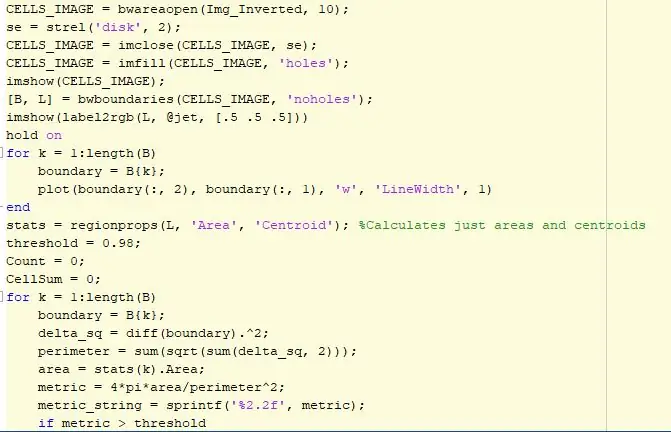

Innan vi kan mäta klumpens rundhet i en bild måste vi konvertera från RGB till gråskala, binära, invertera och filtrera bilden. En filtreringsteknik använder funktionen bwareaopen, som filtrerar bilden av intresse och tar bort alla specifikationer eller pixlar som är för små som inte representerar storleken på en cell. Ett struktureringselement skapas med en form av en disk och ett område på 2 och det används för att fylla i eventuella luckor i bakgrunden eller i cellerna. Sedan använder vi funktionen bwboundaries som spårar blobs och lagrar det i en matris. Vi märker sedan bilden med olika färger så att den blir en tydligare bild. Därefter, med hjälp av en for loop som löper enligt antalet objekt och hål som finns i bilden, sätter den en gräns runt klossarna som motsvarar denna array. När den här slingan är klar börjar en annan slinga, igen enligt antalet objekt och hål som finns i bilden. Den här gången använder vi funktionen regionprops som samlar vissa egenskaper som område från arrayen och lagrar informationen som innehåller antalet objekt och hål. Med hjälp av denna information beräknar vi ytan och omkretsen av dessa objekt med hjälp av centroidformen. Ett tröskelvärde ställs in för att jämföra resultaten när vi har beräknat metriska enheten för ett cirkulärt objekt och plottar denna mätning som text bredvid beräknade centroider. Den slutliga koden visar rundheten hos de olika cellerna som finns i bilden och alla värden nära värdet ett blir mer runda än andra. Ett värde på 1 innebär att cellen är perfekt rund och är i gott skick för att passeras.
Rekommenderad:
Gimbal Stabilizer Project: 9 steg (med bilder)

Gimbal Stabilizer Project: How to Make a Gimbal Lär dig hur du gör en 2-axlig gimbal för din actionkamera I dagens kultur älskar vi alla att spela in video och fånga ögonblicken, särskilt när du är en innehållsskapare som jag, du har säkert mött frågan om så skakig vide
Automatiserat EKG-BME 305 Slutprojekt Extra kredit: 7 steg

Automatiserat EKG-BME 305 Slutprojekt Extra kredit: Ett elektrokardiogram (EKG eller EKG) används för att mäta de elektriska signalerna som produceras av ett hjärtslag och det spelar en stor roll vid diagnos och prognos av hjärt-kärlsjukdom. En del av informationen från ett EKG inkluderar rytmen
CPE 133 Final Project Decimal to Binary: 5 Steg

CPE 133 Final Project Decimal to Binary: Binary Numbers är en av de första sakerna som kommer att tänka på när man tänker på digital logik. Binära siffror kan emellertid vara ett svårt koncept för de som är nya. Detta projekt kommer att hjälpa dem som är både nya och erfarna med binära tal
Akustisk levitation med Arduino Uno Steg-för-steg (8-steg): 8 steg
Akustisk levitation med Arduino Uno Steg-för-steg (8-steg): ultraljudsgivare L298N Dc kvinnlig adapter strömförsörjning med en manlig DC-pin Arduino UNOBreadboardHur det fungerar: Först laddar du upp kod till Arduino Uno (det är en mikrokontroller utrustad med digital och analoga portar för att konvertera kod (C ++)
BME 305 EEG: 4 steg

BME 305 EEG: Ett elektroencefalogram (EEG) är en enhet som används för att mäta ett ämnes elektriska hjärnaktivitet. Dessa tester kan vara mycket användbara för att diagnostisera olika hjärnstörningar. När du försöker göra ett EEG finns det olika parametrar som inte
