Innehållsförteckning:
- Steg 1: Organisera mammogramdata
- Steg 2: Bildbehandling
- Steg 3: Tröskelkod
- Steg 4: Hitta onormaliteter för varje binär bild
- Steg 5: Plotta den diagnostiserade massplatsen och storleken för visuell jämförelse
- Steg 6: Implementering av den andra jämförelsemetoden
- Steg 7: Analysera insamlade data
- Steg 8: Gör din egen klassificerare
- Steg 9: Förbättringar? Några tankar?
- Författare John Day day@howwhatproduce.com.
- Public 2024-01-30 12:45.
- Senast ändrad 2025-01-23 15:11.
Syftet med detta projekt var att identifiera och använda en parameter för att bearbeta gråskala mammogrambilder av olika bakgrundsvävnadsklassificeringar: Fatty, Fatty Glandular och Dense Tissue. Denna klassificering används när radiologer analyserar mammogram och måste överväga om vävnadstäthet kommer att dölja eventuella abnormiteter som skador eller tumörer. Detta beror på att både normala fysiologiska strukturer som körtelvävnad och fibrös bindväv. och onormala morfologier som förkalkningar och tumörer kommer att se mycket ljusa ut på mammogrammet medan mindre tät fettvävnad kommer att se svart ut. Därför var det lämpligt att programmera en klassificerare som kan manipulera pixelintensitetsnivåer för att bäst visualisera och identifiera massor.
Steg 1: Organisera mammogramdata
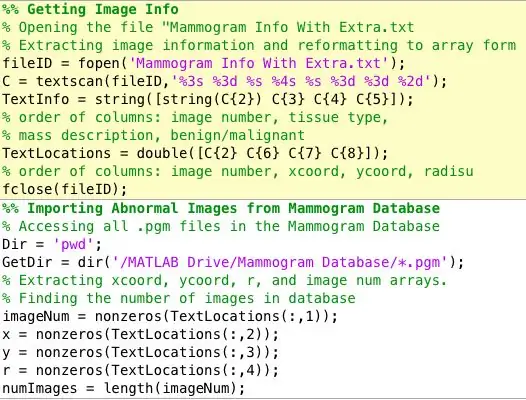
En av de första sakerna jag insåg att jag behövde hantera var att organisera data på ett mycket tydligt, kortfattat och tillgängligt sätt. Dessa är de variabler jag extraherade från mini-MIAS-databasen för mammogram. Jag skapade två matriser. En som innehåller fyra kolumner:
- Bildnummer:
- x massakoordinat
- y massakoordinat
- Massradie: (Detta definierade en ungefärlig storlek för massan
Den andra matrisen innehöll klassificeringsinformation:
- Typ av bakgrundsvävnad: Fett (F), Fettkörtel (G), Tät (D)
- Massbeskrivning: Väldefinierad (CIRC), spiculated (SPIC), illa definierad annan (MISC) Arkitektonisk distorsion (ARCH), Asymmetri (ASYM), Normal (NORM)
- Diagnoser: Godartad (B), Malign (M)
Eftersom målet med detta projekt var att bestämma den bästa tröskeln för varje typ av bakgrundsvävnad var inte all information nödvändig. Du kan dock utöka ditt projekt till att inkludera texturanalys och testa din klassificerare mot de kända massbeskrivningarna.
Sidnotering: Databasen från vilken jag fick de diagnostiserade mammogrambilderna organiserade informationen om varje mammogram i en textfil separat från bilderna. Det var milt svårt för mig att extrahera data från en textfil och organisera i matrisformulär, men följande länk var till stor hjälp för att ta reda på allt detta. Alternativt kan du bara justera koden som jag klistrade in ovan för dina ändamål.
Mammogramfilformat: mdb001 G CIRC B 535 425 197
mdb002 G CIRC B 522 280 69
TextScan Hjälp: https://www.mathworks.com/help/matlab/ref/textsca…Mammogramdatabas:
Steg 2: Bildbehandling
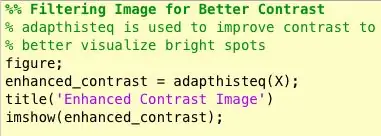
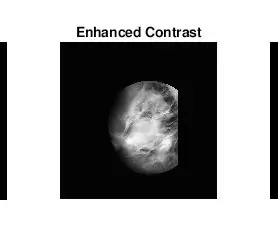
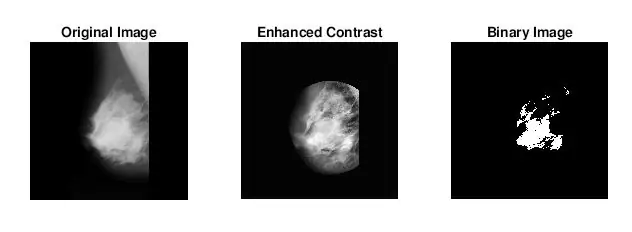
Tja, det andra som kom upp när jag funderade på hur jag skulle identifiera massor var att jag för många onormala mammogram inte visuellt kunde se var avvikelsen var eller hur stor den var. Självklart, eftersom jag inte är en erfaren radiolog, var det förväntat. Men det enklaste sättet att hitta avvikelser (enligt mina långa google -sökningar) var att titta på koncentrationer av ljusa och mörka områden. Jag använde främst adapthisteq -funktionen för att förbättra bildkontrasten och sedan imbinarisera för att konvertera bilden till en binär bild för att experimentera med olika tröskelnivåer.
- adapthisteq: Denna funktion omvandlar intensitetsvärden för gråskala- och rgb -bilder med hjälp av kontrastbegränsad adaptiv histogramutjämning. Med andra ord, det justerar histogrammet för intensitetsvärden till en viss typ av distribution. Matematiklänken för denna funktion är bifogad nedan för vidare läsning.
- imbinarize: skapar en binär bild från en gråskalebild genom att tilldela alla pixlar över en viss intenisti till 1s och pixlarna under det värdet a 0. Jag använde den här funktionen för att testa den optimala tröskeln för att minska bakgrundsvävnadsbrus.
Steg 3: Tröskelkod
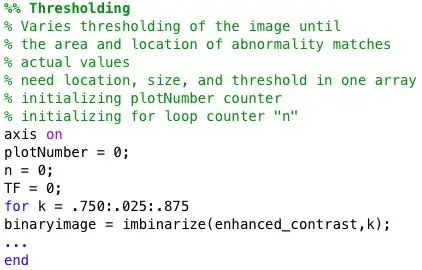
En for loop används för att binarisera mammogrammet med varierande trösklar. För att ge en större bildvy innehåller for -slingan koden från steg 3 till steg 7. Så varje binär bild analyseras för avvikelser. Dessutom är detta för loop inkapslat i ett annat för loop som importerar en ny mammogrambild från databasen i varje iteration.
Steg 4: Hitta onormaliteter för varje binär bild
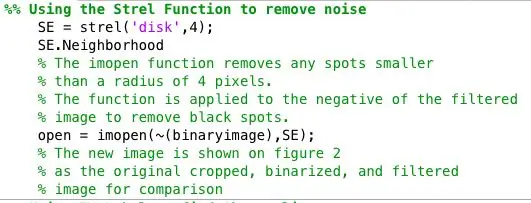
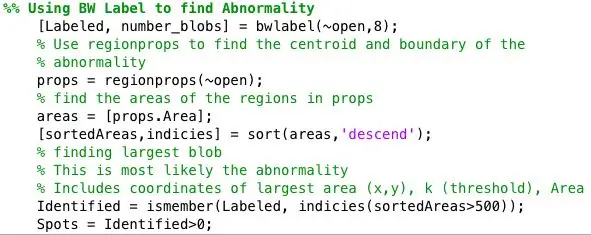
Jag bearbetade vidare de binära bilderna med hjälp av strel -funktionen i samband med imopen för att ta bort bakgrundsbrus. Den binära bilden från föregående steg inverteras och filtreras med hjälp av grannskapet som definieras av SE. Sedan använde jag bwlabel för att märka alla områden som hade minst 8 anslutna pixlar.
Funktionen regionrekvisita användes för att hitta centroid- och areaegenskaperna för varje plats som identifierades med bwlabel.
Sedan identifierades alla fläckar större än 500 pixlar med hjälp av ismember. Centroiderna för de identifierade fläckarna ritades ut på en bild som bara visade fläckarna större i area än 500. Area Identified = ismember (märkt, indikerar (sortedAreas> 500)); Fläckar = Identifierade> 0;
Steg 5: Plotta den diagnostiserade massplatsen och storleken för visuell jämförelse
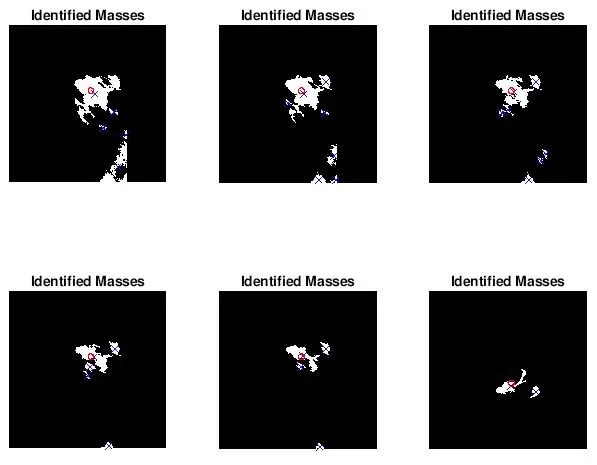
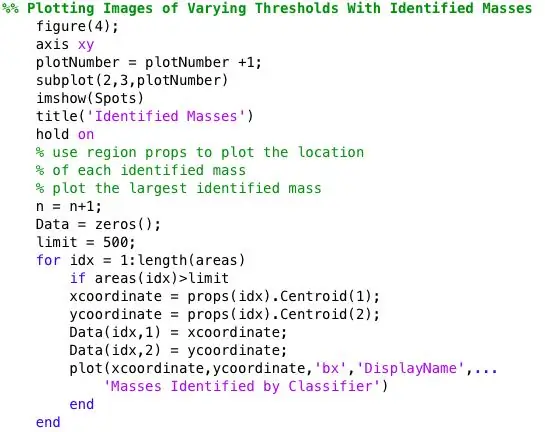

Jag ville se om fläckarna som hittades av bwlabel var korrekta. Jag gjorde detta på två sätt. Jag analyserade först noggrannheten i min klassificerare genom att göra en visuell jämförelse. Jag ritade helt enkelt den verkliga storleken och platsen för abnormiteten (röd cirkel) och platsen som bestäms av koden (blå x) på den förbehandlade mammogrambilden. De sex bilderna ovan visar effekterna av att öka tröskelvärdet för gråskala.
Steg 6: Implementering av den andra jämförelsemetoden

Det andra sättet jag testade klassificeraren och tröskelvärdena var genom att avgöra om platserna som klassificeraren hittade var inom ett visst avstånd från de diagnostiserade abnormitetskoordinaterna. Jag sparade trösklarna för vilka minst en av de identifierade punkterna låg inom 1,5*r från den kända abnormiteten till en separat textfil som heter Mammogramdata. Syftet med detta var att hitta det minsta tröskelvärdet för min klassificerare för att identifiera avvikelsen.
Steg 7: Analysera insamlade data
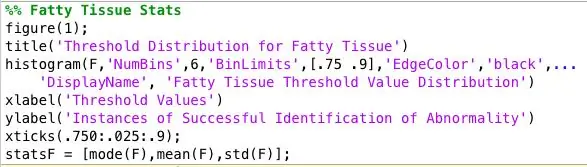
Jag körde programmet på alla onormala mammogrambilder och jag fick en enorm textfil med data. För att hitta den bästa tröskeln för varje typ av vävnad organiserade jag data efter vävnadstyp och ritade upp ett histogram av tröskelvärdena för varje vävnadstyp. Det rätta tröskelvärdet bestämdes på vilket tröskel som gav de mest exakta resultaten för varje vävnadstyp. Jag sparade dessa data för att ladda upp till min klassificerare.
Steg 8: Gör din egen klassificerare

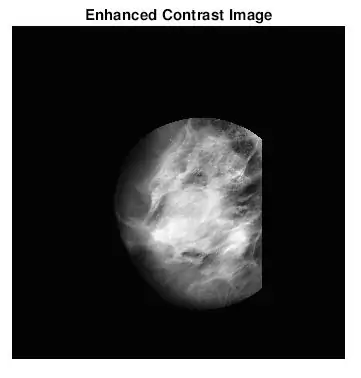
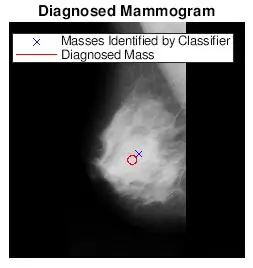
Efter att jag hittat de mest lämpliga tröskelvärdena för varje vävnadstyp, redigerade jag min ursprungliga kod för att få en användare att mata in bildnummer och vävnadstyp för att välja tröskeln för mammogrambilden. Jag ritade sedan upp den diagnostiserade mammogramplatsen med de hittade platserna på de ursprungliga mammogrambilderna. Jag ville göra det här roligare så jag programmerade en funktion för att beskära ett cirkulärt område som omger ROI. Användaren skulle instrueras att välja en mittpunkt och flera punkter som bäst omfattar ROI. Jag bifogade båda matlabfilerna här.
Steg 9: Förbättringar? Några tankar?
När jag skrev detta instruerbara börjar jag se många förbättringar som jag kunde göra för klassificeraren, till exempel att hitta sätt att skilja mellan olika typer av massor som identifierats baserat på texturanalys eller förbättra min testning för noggrannhetssektionen i SandBoxProject. fil. Eftersom det här var ett projekt med en deadline måste jag sluta någonstans, men jag hoppas att jag kommer att kunna använda de bildbehandlingsförmågor jag lärde mig i andra applikationer. Jag bifogade också filen som användes för att satsvis bearbeta alla onormala mammogrambilder.
Rekommenderad:
[2020] Använda iPhone eller iPad och Micro: bit Game Pad -app för att styra en RC -bil: 23 steg
![[2020] Använda iPhone eller iPad och Micro: bit Game Pad -app för att styra en RC -bil: 23 steg [2020] Använda iPhone eller iPad och Micro: bit Game Pad -app för att styra en RC -bil: 23 steg](https://i.howwhatproduce.com/images/001/image-1423-j.webp)
[2020] Använda iPhone eller iPad och Micro: bit Game Pad App för att styra en RC -bil: Har du tänkt använda din iPhone eller iPad för att styra din micro: bit? Vet du att Micro: bit Educational Foundation tillhandahåller iOS -appen i App store? Sök efter " micro: bit " i App Store och du kan ladda ner appen gratis. De
Visualisera barometertryck och temperatur med Infineon XMC4700 RelaxKit, Infineon DPS422 och AWS .: 8 steg
Visualisera barometertryck och temperatur med Infineon XMC4700 RelaxKit, Infineon DPS422 och AWS .: Det är ett enkelt projekt för att fånga barometertryck och temperatur med Infineons DPS 422. Det blir klumpigt att spåra tryck och temperatur över en tidsperiod. Det är här analytics kommer in i bilden, insikten om förändringen i
DIY MusiLED, musiksynkroniserade lysdioder med ett klick Windows och Linux-applikation (32-bitars och 64-bitars). Lätt att återskapa, lätt att använda, lätt att porta .: 3 steg

DIY MusiLED, musiksynkroniserade lysdioder med ett klick Windows och Linux-applikation (32-bitars och 64-bitars). Lätt att återskapa, lätt att använda, lätt att porta .: Detta projekt hjälper dig att ansluta 18 lysdioder (6 röda + 6 blå + 6 gula) till ditt Arduino-kort och analysera datorns ljudkortets realtidsignaler och vidarebefordra dem till lysdioderna för att tända dem enligt slageffekterna (Snare, High Hat, Kick)
Övertyga dig själv om att bara använda en 12V-till-AC-omriktare för LED-ljussträngar istället för att koppla om dem för 12V: 3 steg

Övertyga dig själv om att bara använda en 12V-till-AC-linjeomvandlare för LED-ljussträngar istället för att koppla om dem för 12V: Min plan var enkel. Jag ville klippa upp en väggdriven LED-ljussträng i bitar och sedan dra om den för att gå av 12 volt. Alternativet var att använda en kraftomvandlare, men vi vet alla att de är fruktansvärt ineffektiva, eller hur? Höger? Eller är de det?
Att använda Parallax Basic Stamp II för att ringa en dörrklocka på distans: 4 steg (med bilder)

Att använda Parallax Basic Stamp II för att ringa en dörrklocka på distans: Problemet? En hund som blir alldeles för upphetsad när dörrklockan ringer. Lösningen? Ring på dörren vid slumpmässiga tillfällen när ingen är där, och ingen svarar på den, för att motkonditionera hunden - för att bryta föreningen som en ringande dörrklocka e
