
Innehållsförteckning:
- Steg 1: Konfigurera en Google Cloud Storage Bucket
- Steg 2: Formatera dina data och skapa datauppsättning Csv
- Steg 3: Ladda upp dina spektrogram till din hink
- Steg 4: Ladda upp din datauppsättning Csv
- Steg 5: Skapa datauppsättning
- Steg 6: Skapa din AutoML -modell
- Steg 7: Testa din modell
- Steg 8: Installera din modell i ThinkBioT
- Författare John Day day@howwhatproduce.com.
- Public 2024-01-30 12:44.
- Senast ändrad 2025-01-23 15:11.

ThinkBioT är designat för att vara "Plug and Play", med Edge TPU -kompatibla TensorFlow Lite -modeller.
I denna dokumentation kommer vi att omfatta att skapa spektrogram, formatera dina data och använda Google AutoML.
Koden i denna handledning kommer att skrivas i bash så kommer att vara kompatibel med flera plattformar.
Beroenden
- Men innan du börjar måste du installera Sox ett kommandorads ljudprogram som är kompatibelt med Windows-, Mac- och Linux -enheter.
- Om du använder en Windows -enhet är det enklaste sättet att köra bash -skript via Git så jag skulle rekommendera och ladda ner och installera det eftersom det är användbart på många sätt,
- För att redigera kod använder du antingen din favoritredigerare eller installerar NotePad ++ för Windows eller Atom för andra operativsystem.
** Om du har en befintlig TensorFlow -modell eller vill försöka överföra inlärning med en befintlig modell, se Googles koraldokumentation.
Steg 1: Konfigurera en Google Cloud Storage Bucket
1. Logga in på ditt gmail -konto (eller skapa ett om du inte har ett Google -konto)
2. Gå till projektväljarsidan och skapa ett nytt projekt för dig modell- och spektrogramfiler. Du måste aktivera faktureringen för att gå vidare.
3. Besök https://cloud.google.com/storage/ och tryck på knappen skapa hink högst upp på sidan.
4. Ange önskat skopnamn och skapa skopan som accepterar standardinställningar.
Steg 2: Formatera dina data och skapa datauppsättning Csv
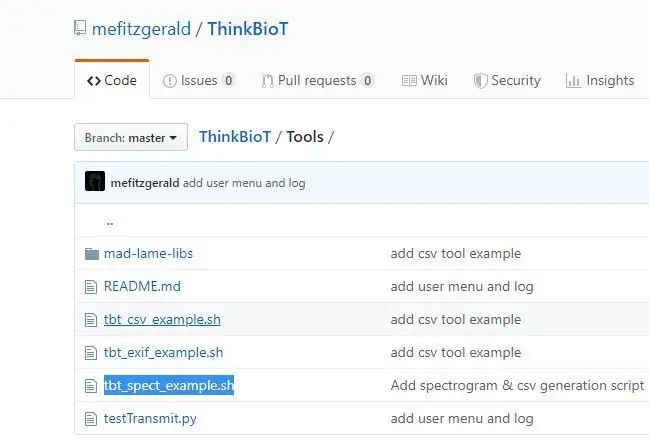
Jag har utformat ett användbart skript för att skapa din dataset.csv -fil som behövs för att skapa din modell. Datafilen länkar bilderna i din hink till deras etiketter i datamängden.
1. Ladda ner ThinkBioT -förvaret från GitHub och
2. Kopiera filen tbt_spect_example.sh från Verktygskatalogen till en ny mapp på skrivbordet.
3. Lägg till de ljudfiler du vill använda i din modell och lägg dem i mappar som har deras etikett (dvs. vad du vill att de ska sorteras i. Om du till exempel vill identifiera hundar eller katter kan du ha en mapp hund, med barkljud ELLER mapp som heter katt med kattljud osv.
4. Öppna tbt_spect_example.sh med Anteckningar ++ och ersätt "dittnamn" på rad 54 med namnet på din Google Storage Bucket. Till exempel, om din hink kallades myModelBucket skulle raden ändras till
bucket = "gs: // myModelBucket/spectro-data/"
5. Kör koden genom att skriva följande i din Bash-terminal, koden körs och skapar dina etiketter csv-fil och en katalog som kallas spektrodata på skrivbordet med de resulterande spektrogrammen.
sh tbt_spect_example.sh
Steg 3: Ladda upp dina spektrogram till din hink
Det finns några sätt att ladda upp till Google Storage, det enklaste är att göra en direkt mapp för att ladda upp;
1. Klicka på ditt skopnamn på din Google Storage -sida.
2. Välj "UPLOAD FOLDER" -knappen och välj din "spectro-data/" -katalog som skapades i det sista steget.
ELLER
2. Om du har en stor mängd filer kan du manuellt skapa "spectro-data/" -katalogen genom att välja "SKAPA MAPP", navigera sedan till mappen och välja "UPLOAD FILES". Detta kan vara ett bra alternativ för stora datamängder eftersom du kan ladda upp spektrogrammen i sektioner, till och med använda flera datorer för att öka uppladdningshastigheten.
ELLER
2. Om du är en avancerad användare kan du också ladda upp via Google Cloud Shell;
gsutil cp spectro-data/* gs: // your-bucket-name/spectro-data/
Du borde nu ha en hink full av vackra vackra spektrogram!
Steg 4: Ladda upp din datauppsättning Csv
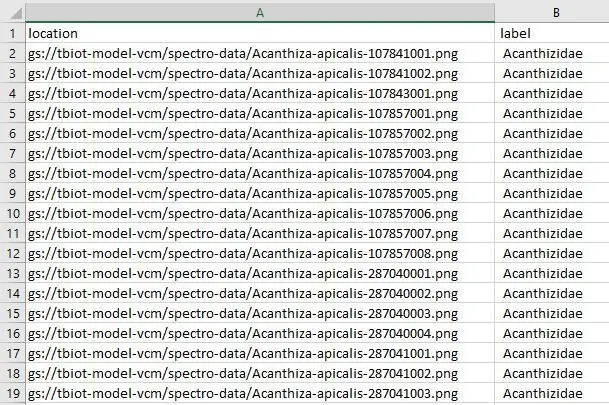
Nu måste vi ladda upp filen model-labels.csv till din "spectro-data/" -katalog i Google Storage, det är i stort sett samma som det sista steget, du laddar bara upp en enda fil istället för många.
1. Klicka på ditt skopnamn på din Google Storage -sida.
2. Välj knappen "UPLOAD FILE" och välj din modell-labels.csv-fil som du skapade tidigare.
Steg 5: Skapa datauppsättning
1. För det första måste du hitta AutoML VIsion API, det kan vara lite knepigt! Det enklaste sättet är att söka efter "automl vision" i sökfältet i ditt Google Cloud -lagringsutrymme (bilden).
2. När du klickar på API -länken måste du aktivera API: et.
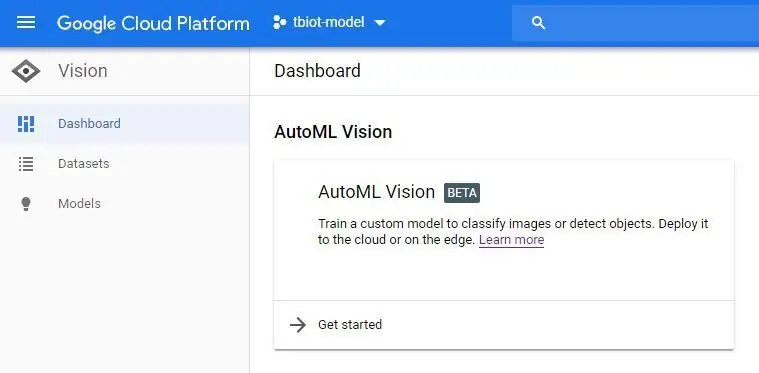
3. Nu kommer du att vara i AutoML Vision Dashboard (bilden) klicka på den nya dataknappen och välj Enkel etikett och alternativet "Välj en CSV -fil". Du kommer sedan att inkludera länken till din modell-labels.csv-fil i din lagringshink. Om du har följt denna handledning kommer det att vara enligt nedan
gs: //yourBucketName/spectro-data/model-labelsBal.csv
4. Tryck sedan på Fortsätt för att skapa din datauppsättning. Det kan ta lite tid att skapa.
Steg 6: Skapa din AutoML -modell
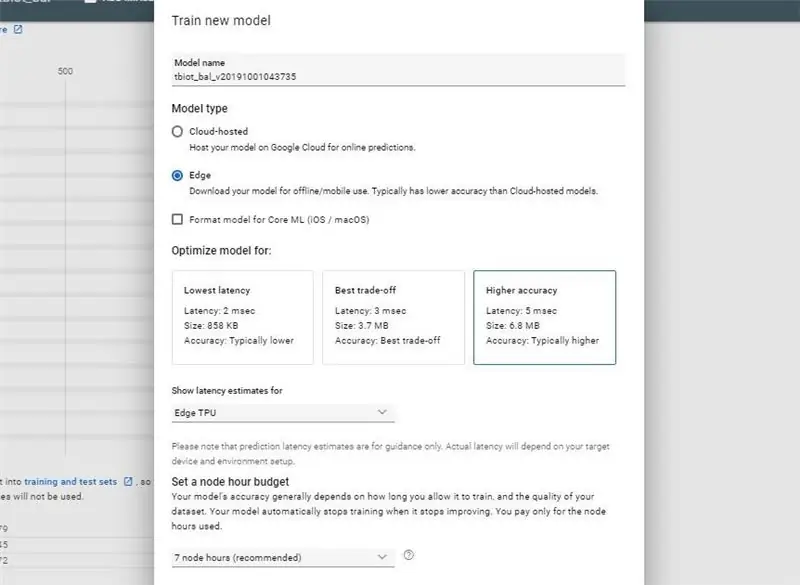

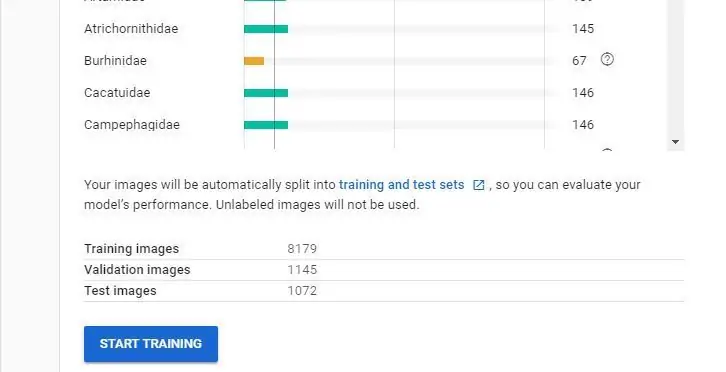
När du har fått ditt e -postmeddelande om att din dataset har skapats är du redo att skapa din nya modell.
- Tryck på TRAIN -knappen
- Välj modelltyp: Uppskattningar av kant- och modelllatens: Kant -TPU och lämna de andra alternativen som standard till en början, tufft du kanske vill experimentera med sedan senare.
- Nu kommer din modell att träna, det tar lite tid och du får ett mejl när den är klar att ladda ner.
Obs! Om du tränar är knappen inte tillgänglig kan du ha problem med din datamängd. Om du har mindre än 10 av varje klass (etikett) låter systemet dig inte träna en modell så att du kan behöva lägga till extra bilder. Det är värt att titta på Google AutoML -videon om du behöver förtydligande.
Steg 7: Testa din modell
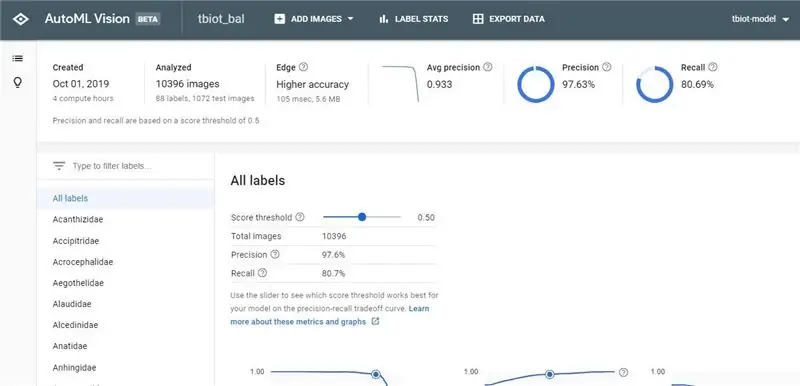
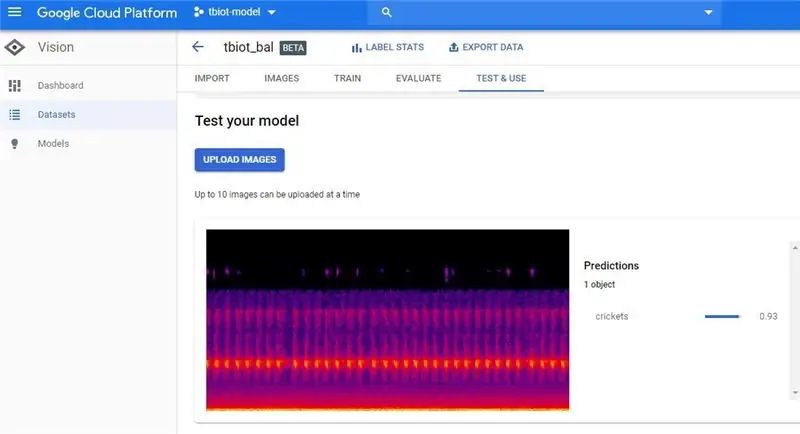
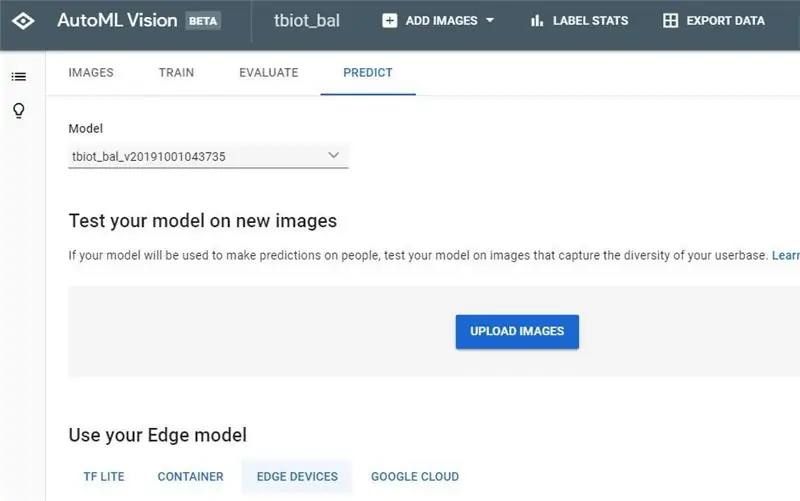
När du har fått ditt e -postmeddelande om färdigställande av klickar du på länken för att återgå till AutoML Vision API.
1. Nu kommer du att kunna se dina resultat och förvirringsmatrisen för din modell.
2. Nästa steg är att testa din modell, gå till "TEST & USE" eller "PREDICT" konstigt att det verkar finnas två användar -GUI: er, som jag båda har bildat, men alternativen båda har samma funktionalitet.
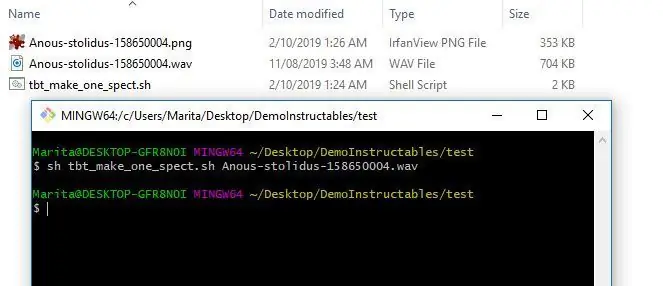
3. Nu kan du ladda upp ett testspektrogram. För att göra ett enda spektrogram kan du använda programmet tbt_make_one_spect.sh från ThinkBioT Github. Släpp den helt enkelt i en mapp med den wav som du vill konvertera till ett spektrogram, öppna ett Git Bash -fönster (eller terminal) och använd koden nedan och ersätt ditt filnamn.
sh tbt_make_one_spect.sh yourWavName.wav
4. Ladda nu upp spektrogrammet och kontrollera ditt resultat!
Steg 8: Installera din modell i ThinkBioT
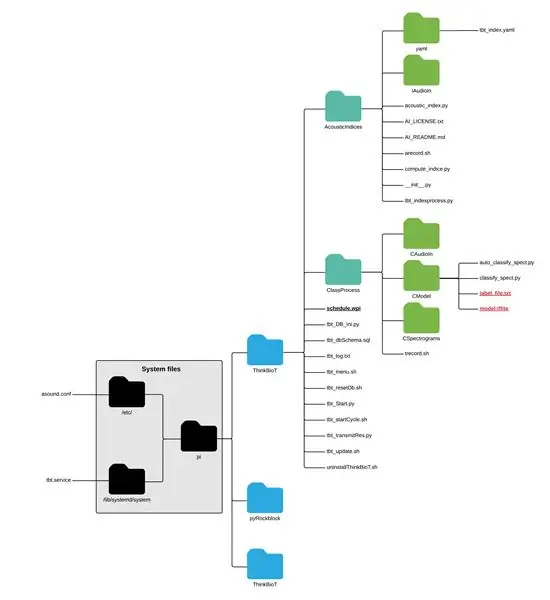
För att använda din nya blanka modell släpper du bara modellen och txt -filen i CModel -mappen;
pi> ThinkBioT> ClassProcess> CModel
Nu är du redo att använda ThinkBioT:)
** OBS ** Om du använder din modell utanför ThinkBioT-ramverket måste du redigera ditt etikettdokument som tilläggsnummer i början av varje rad, eftersom de senaste tflite-tolkarna inbyggda "readlabels" -funktionen förutsätter att de finns där. Jag har skrivit en anpassad funktion i ThinkBioT -ramverket classify_spect.py som ett arbete som du är välkommen att använda i din egen kod:)
def ReadLabelFile (file_path):
räknare = 0 med öppen (file_path, 'r', encoding = 'utf-8') som f: lines = f.readlines () ret = {} för rad i rader: ret [int (counter)] = line.strip () räknare = räknare + 1 returret
Rekommenderad:
Rörelseaktiverade cosplayvingar med Circuit Playground Express - Del 1: 7 steg (med bilder)

Motion Activated Cosplay Wings Using Circuit Playground Express - Del 1: Detta är del ett av ett tvådelat projekt, där jag ska visa dig min process för att göra ett par automatiska älvvingar. Den första delen av projektet är vingarnas mekanik, och den andra delen gör den bärbar och lägger till vingarna
Väderdata med hjälp av Google Sheets och Google Script: 7 steg

Väderdata med hjälp av Google Kalkylark och Google -skript: I den här bloggtutan kommer vi att skicka avläsningarna av SHT25 -sensorn till Google -ark med Adafruit huzzah ESP8266 som hjälper till att skicka data till internet. Skicka data till Google bladcell är mycket användbart och grundläggande sätt som sparar data på
Del 1. ThinkBioT autonom bio-akustisk sensor Hårdvara Bygg: 13 steg

Del 1. ThinkBioT Autonomous Bio-acoustic Sensor Hardware Build: ThinkBioT syftar till att tillhandahålla en ram för programvara och hårdvara, utformad som en teknisk ryggrad för att stödja ytterligare forskning, genom att hantera uppgifter om datainsamling, förbehandling, dataöverföring och visualiseringsuppgifter som möjliggör forskare
Trådlös fjärrkontroll med 2,4 GHz NRF24L01 -modul med Arduino - Nrf24l01 4 -kanals / 6 -kanals sändarmottagare för Quadcopter - Rc helikopter - RC -plan med Arduino: 5 steg (med bi
Trådlös fjärrkontroll med 2,4 GHz NRF24L01 -modul med Arduino | Nrf24l01 4 -kanals / 6 -kanals sändarmottagare för Quadcopter | Rc helikopter | Rc -plan med Arduino: Att driva en Rc -bil | Quadcopter | Drone | RC -plan | RC -båt, vi behöver alltid en mottagare och sändare, antag att för RC QUADCOPTER behöver vi en 6 -kanals sändare och mottagare och den typen av TX och RX är för dyr, så vi kommer att göra en på vår
IoT -kattmatare med partikelfoton integrerat med Alexa, SmartThings, IFTTT, Google Sheets: 7 steg (med bilder)

IoT -kattmatare med partikelfoton integrerat med Alexa, SmartThings, IFTTT, Google Sheets: Behovet av en automatisk kattmatare är självförklarande. Katter (vår katt heter Bella) kan vara vidriga när de är hungriga och om din katt är som min kommer den att äta skålen torr varje gång. Jag behövde ett sätt att automatiskt dosera en kontrollerad mängd mat
