Innehållsförteckning:
- Steg 1: Använda Brain Box
- Steg 2: Intro: Analysera tvärsnitt
- Steg 3: Konfigurera en funktion: LevelCurveTracings.m
- Steg 4: Utveckla den bundna algoritmen: Hitta gränser
- Steg 5: Utveckla den bundna algoritmen: Generera yttre bunden matris
- Steg 6: Utveckla den bundna algoritmen: Arbeta med mittpunkt
- Steg 7: Utveckla den bundna algoritmen: När en Centroid inte är centrerad
- Steg 8: Utveckla den bundna algoritmen: störningar av hål
- Steg 9: Utveckla den bundna algoritmen: lokalisera hål, slutföra hjärngränser och hålgränser
- Steg 10: Loggningsdata: Funktion PatientFiles.m
- Steg 11: Logga in data i filer
- Steg 12: Loggningsdata: Visning av en del hjärnvolym över tid
- Steg 13: Stänga luckor i delplaner: Subplotclose.m
- Steg 14: Huvudkoden: Rensa allt och be om inmatningar
- Steg 15: Huvudkoden: Batchbehandling av bilderna
- Steg 16: Huvudkoden: Padding
- Steg 17: Huvudkoden: Fastställande av gränser
- Steg 18: Huvudkoden: Bestämning av korrekt Z -dimension
- Steg 19: Huvudkoden: Bestämning av X- och Y -koordinater
- Steg 20: Huvudkoden: Plotta en tredimensionell struktur, hitta volym och logga data
- Författare John Day day@howwhatproduce.com.
- Public 2024-01-31 10:25.
- Senast ändrad 2025-01-23 15:11.
Framsteget till gränsen för ett längre mänskligt liv har lett till ökningen av sjukdomar som civilisationer inte sett före vårt. Bland dessa drabbade Alzheimers cirka 5,3 miljoner levande äldre amerikaner 2017, eller cirka 1 av 10 äldre amerikaner (https://www.alz.org/facts/) och otaliga andra med demens. För att hjälpa till i kampen för att förstå vad som drabbar våra äldste, kommer denna kod att utrusta framtida forskare och ivriga nyfikna med förmågan att spåra hjärnvolym över tid.
Steg 1: Använda Brain Box
För att använda hjärnbox behöver man bara följande:
- MR -skanningar av en hjärna och namn och format på sådana filer (bör alla ha ungefär samma dimensioner)
- Längd på en skanning
- Avstånd mellan varje lager (MR -skanning)
- Patientnamn (inkludera inte mellanslag när du matar in. Var vänlig och versera förnamn och efternamn, till exempel: Förnamn Efternamn)
Och från detta har man förmågan att hålla reda på individers trender i hjärnvolym över tid. Således kan siffror för Alzheimers trender spåras av denna programvara. Längden vi använde i försöket var 180 mm för längden på en skanning och 5 mm för avståndet mellan MR -skanningar, baserat på genomsnittliga siffror.
Tillämpningen av hjärnbox behöver dock inte begränsas till denna enda uppgift. Om tvärsnittet av ett givet fast ämne är fotografi, som en tumör i sig, kan trenderna i volymförändringar för dessa också spåras i programvaran.
Steg 2: Intro: Analysera tvärsnitt
I tredimensionella strukturer skulle de tvådimensionella planen som sådana består av kallas tvärsnitt. Tänk dig att en bunt med papper utgör ett rektangulärt prisma, då skulle varje pappersbit vara ett tvärsnitt av papperet. När vi föreställer oss hjärnan tillämpar vi samma tankesätt. MR (magnetisk resonansavbildning) (se information om MR) fångar hjärnans tvärsnitt och från att använda gränserna definierade i varje "lager" i hjärnan som tillhandahålls kan vi konstruera en struktur för att modellera och hitta hjärnans volym. Vi måste dock först bygga en funktion för att ge information om sådana gränser.
Steg 3: Konfigurera en funktion: LevelCurveTracings.m
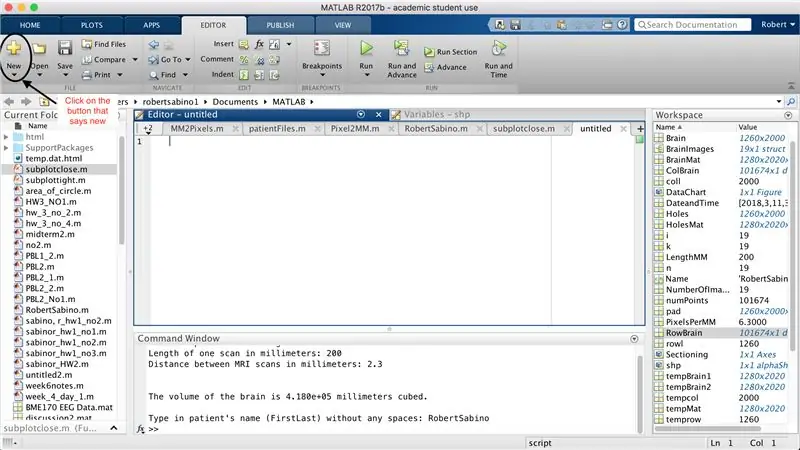
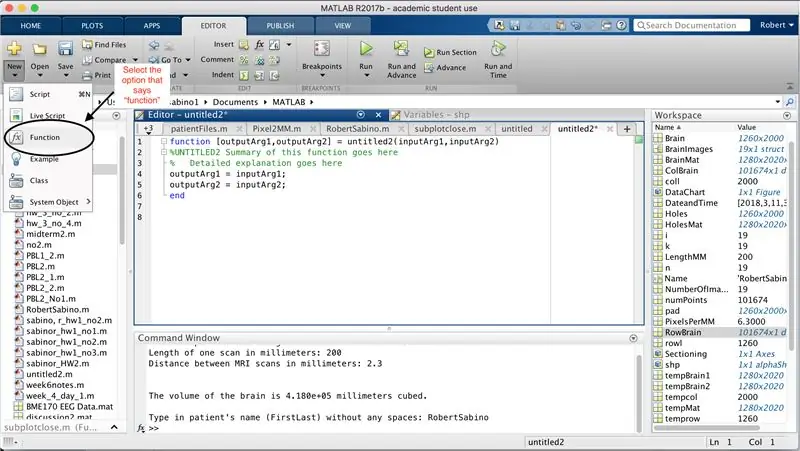
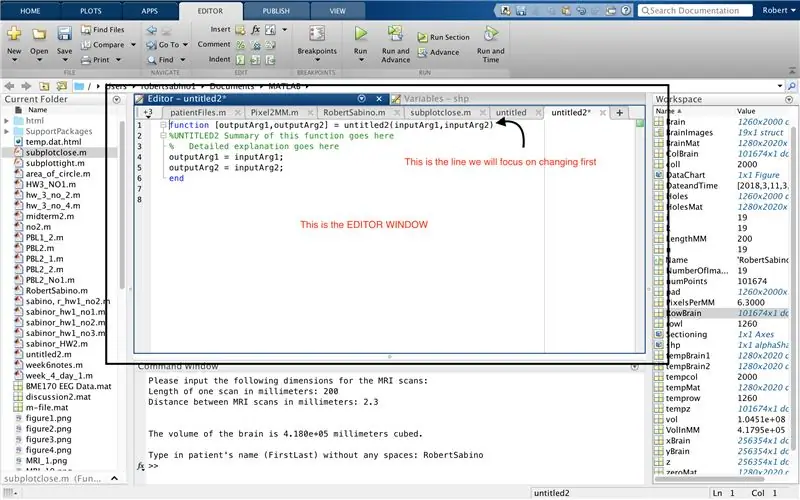

Kontrollera först att din dator har MATLAB_R2017b nedladdad (ladda ner här) och öppna MATLAB. I MATLAB -gränssnittet klickar du på knappen i det övre vänstra hörnet av fönstret där det står "Nytt" med ett fet gult plustecken, och väljer alternativet "funktion" för att öppna ett utrymme i editorfönstret som liknar det i tredje bilden. Vi kommer att fokusera på att ändra den första raden för att konfigurera funktionen. Där det står "outputArg1", ersätt det med "brain", "outputArg2" för att säga "hole", "untitled2" till "exp2" och "inputArg1" till "image" och radera "inputArg2". Du har nu en funktion som ska kallas med hjälp av "exp2", ta ett argument "bild" och sätta gränserna för "hjärna" och "hål". Den första raden i funktionen ska likna linjen som visas på den fjärde bilden. Radera all kod nedanför denna första rad.
Steg 4: Utveckla den bundna algoritmen: Hitta gränser

Skriv in koden enligt följande under raden. Den här delen av funktionen gör följande rad-för-rad.
- Ladda in bilden "bild" i variabeln "mri".
- Förvandla "mri" till en bild gjord av värden i ett antal nummer till ettor och nollor (aka binärisering) baserat på ett inställt tröskelvärde. Om värdet i en pixel är lika med eller större än 0,1, sätts det till en, om inte, sätts värdet vid den pixeln till noll.
- Följande fyra rader förvandlar 10 kolumner och rader vid kanterna på MR -skanningsskiktet till nollor, för att undvika att läsa felaktiga värden som att göra gränser (som vi lärde oss av att experimentera med koden).
- På den sista raden spårar bwboundaries gränserna för den binära bilden "mri" och sätter den lika med "b", en matris med elementen vars index motsvarar gränsvärdena som är inställda på en.
Steg 5: Utveckla den bundna algoritmen: Generera yttre bunden matris
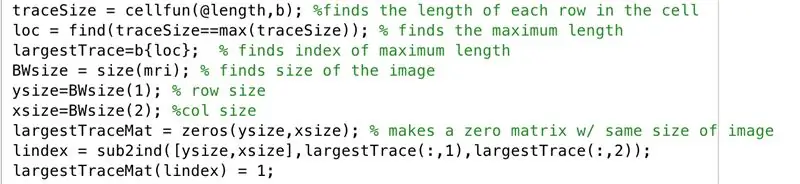
Följ med i redigeringsfönstret med följande kod på bilden. Denna del av koden gör följande rad-för-rad.
- Hitta längden på var och en av raderna i den binära bilden "b" (cellfun tillämpar funktionslängden på varje rad).
- Ställ in "loc" för att lagra maximala längder.
- Hitta indexet för den maximala längden, inställd på att lagra i "grutsteTrace".
- Hitta storleken på bilden "mri", som består av samma storlek som "b", och ställ in på "BWsize".
- Hitta antalet rader i bildens array, ställ in på "ysize".
- Hitta antalet kolumner i bildens array, inställt på "xsize".
- Generera matris "grootsteTraceMat", en "ysize" med "xsize" -matris med nollor.
- Hitta det ekvivalenta indexet från de subskriberade värdena som motsvarar där de störstaTrace x -värdena och y -värdena var, lagra i vektorn "lindex".
- I matrisen med nollor, "grutsteTraceMat", gör elementen vid indexen som motsvarar indexvärden lagrade som element i "lindex" till ettor.
Således har den logiska matrisen "LargestTraceMat" den största avgränsade regionen av det givna hjärnskannatvärsnittet ritat som de med en bakgrund av nollor
Steg 6: Utveckla den bundna algoritmen: Arbeta med mittpunkt

Därefter måste vi testa om tvärsnittet består av mer än en region (den största). Genom att testa inriktningen av den största regionens centroid kan vi se om det finns en sammanhängande region, vilket skulle ge en mer centrerad centroid eller möjligheten för flera regioner.
- Använd "regionProps" för att hitta information om centroiderna som finns, lika med strukturmatrisen "tempStruct"
- Formera "centroids" med data från fältet "centroid" sammanfogade vertikalt
- Ta de andra kolumnvärdena för "centroids" (de horisontella dimensionskoordinaterna)
- Kör ett filter för att kontrollera centroidens inriktning mot den horisontella mitten
Steg 7: Utveckla den bundna algoritmen: När en Centroid inte är centrerad
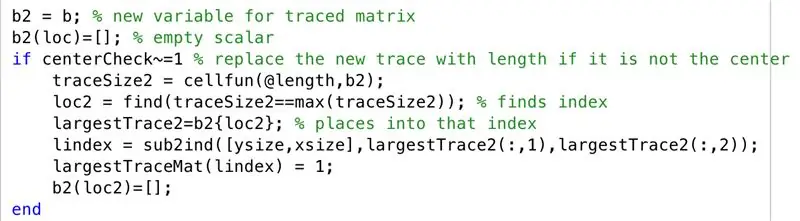
I scenariot att centroiden för den största spårregionen inte är centrerad går vi igenom följande steg. Som vi hade observerat i MR -skanningarna var tendensen att få hjärnhalvorna avbildade i tvärsnittet när de inte var sammanhängande, så vi fortsätter nu att plotta det näst största spåret tillsammans med det största spåret i "grutsteTraceMat"
- Ställ in den spårade matrisen till en ny variabel "b2"
- Initiera tom matris "b2", med en uppsättning indexerad av "loc"
- Skapa en villkorlig, för när en centroid inte är centrerad (dvs. ett lager med flera regioner)
- Ange en ny spårningsstorlek för varje rad (traceSize2)
- Ställ in "loc2" för att hitta index där gränser finns
- Låt celler som anges med "loc2" i "b2" vara lika med "grutsteTrace2"
- Konvertera prenumerationer till index, inställt på "lindex"
- Ändra element som motsvarar "lindex" i "grutsteTraceMat" till 1
- Initiera tom matris "b2", med en uppsättning indexerad av "loc2"
Steg 8: Utveckla den bundna algoritmen: störningar av hål

Vid hantering av hål, de värden som lagras i "b2" höll koll på andra strukturer än det största spåret, och plotta dessa på en fylld form av "grutsteTraceMat" kommer att avslöja var det finns hål i hjärnregionerna.
- Skapa matris "filledMat", som är en fylld i form av "LargestTraceMat"
- Skapa matris "interferenceMat", en "ysize" med "xsize" array med nollor
- Skapa matris "interferenceloc" för att lagra värdena från "b2", sammankopplade vertikalt
- Skapa array "lindex" för att lagra index som motsvarar "interferenceloc"
- För index i "interferenceMat" som motsvarar "lindex", sätt värdet till 1, vilket gör en annan avgränsad region
Steg 9: Utveckla den bundna algoritmen: lokalisera hål, slutföra hjärngränser och hålgränser

- Ställ in matris "tempMat" lika med "interferenceMat" plus "filledMat" och lägg till varje värde i matrisen till varandra
- Ange array "holesLoc" lika med index där "interferenceMat" och "filledMat" båda var lika med en
- Konfigurera "holesMat" som en nollmatris med dimensioner "ysize" x "xsize"
- Ange index i "holesMat" som är lika med "holesLoc" som sådana
- Ställ in "hjärna" till "grutsteTraceMat"
- Ställ in "hål" på "holeMat"
Med fyndet där värdena för de tillsatta matriserna var lika med 2, var hålplatserna enkelt säkrade och ritade på en tom matris.
Steg 10: Loggningsdata: Funktion PatientFiles.m
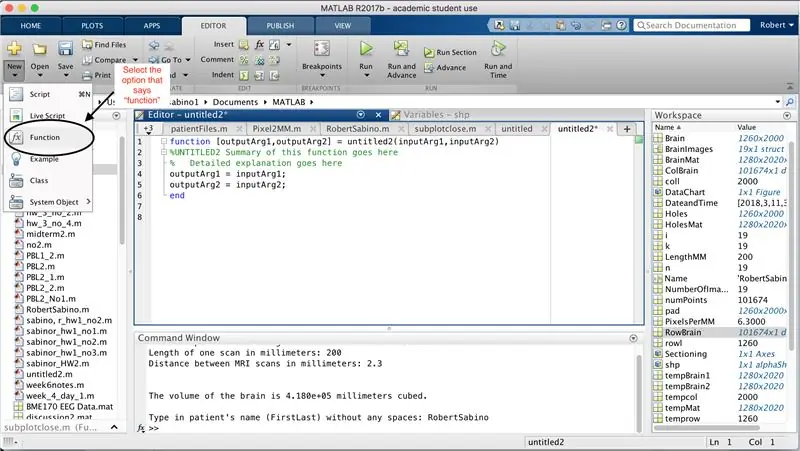
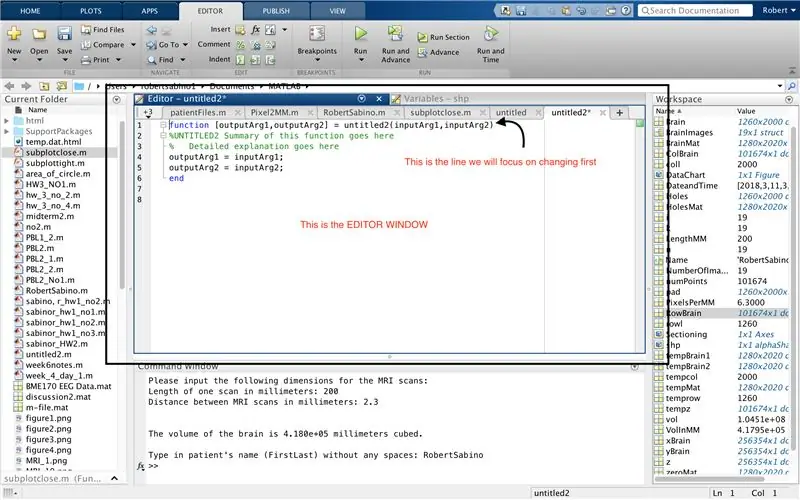

Ungefär som installationen av den senaste funktionen, klicka på knappen i det övre vänstra hörnet av fönstret där det står "Nytt" med ett fet gult plustecken och välj alternativet "funktion" för att öppna ett utrymme i redigeringsfönstret liknar det på den tredje bilden. På den första raden, ta bort utmatrisen och ersätt med bara "output", ersätt "untitled2" med "patientFiles", ta bort alla inmatningsargument och följ istället den formatering som anges i den fjärde bilden av kodraden. Den första raden i den här funktionen ska matcha bildens formatering.
Steg 11: Logga in data i filer
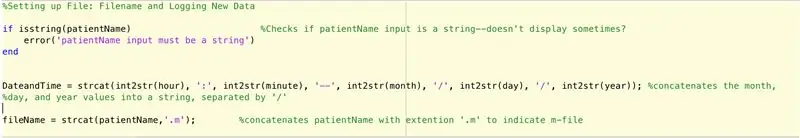

För att ställa in en fil för att logga data som hittats av huvudfunktionen (som ännu inte ska beskrivas) måste vi följa dessa steg (enligt koden rad för rad).
- Kontrollera om input för patientName är en sträng.
- Om det inte är en sträng, visa att patientName -ingången ska vara en sträng.
- Avsluta if -satsen (förhindra fel).
- Skapa ett stränguttalande "DateandTime" som ger följande format: timme: minuter-månad/dag/år.
- Ställ in variabel filnamn på följande: patientName.m.
Nu till nästa avsnitt i funktionen: Finns det redan en fil med detta namn?
1) Anta att filen med detta namn redan finns:
- Kör filen för att få värdena från det förra köat
- Lägg till "DateandTime" -data för den aktuella iterationen som en ny cell i cellmatrisen med x -värden (indexänd+1)
- Lägg till det aktuella värdet "brainVolume" som en ny cell i cellmatrisen med y -värden (indexänd+1)
- Spara de aktuella variablerna som laddats i filen.
2) Anta att filen med detta namn inte finns:
- Skapa en ny fil med namnet lagrat i variabeln "patientnamn"
- Lägg till den aktuella "DateandTime" -datan som en cell i den tomma cellmatrisen med x -värden
- Lägg till nuvarande "brainVolume" -data som en cell i den tomma cellmatrisen med y -värden
- Spara de aktuella variablerna som laddats i filen.
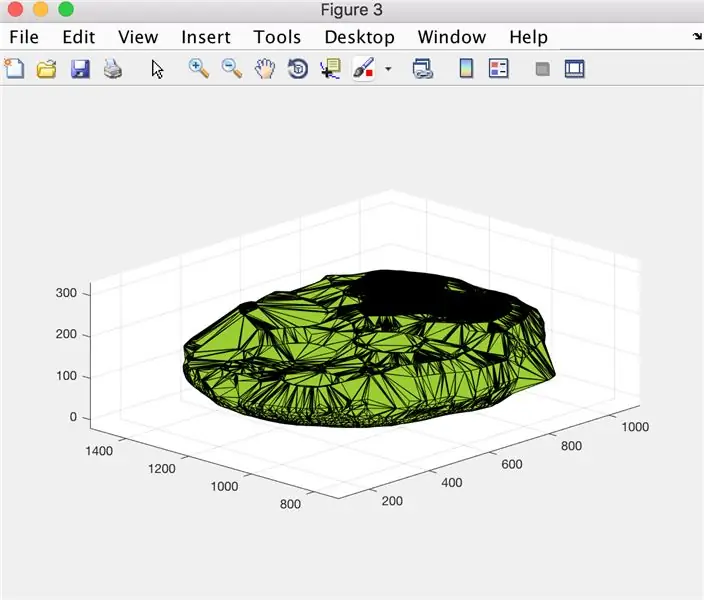
Steg 12: Loggningsdata: Visning av en del hjärnvolym över tid

- Konvertera x -värden array (xVals) till en kategorisk array (xValsCategorical), för att tillåta plottning
- Skapa figurfönster 5
- Plotta punkterna som anges med "xValsCategorical" och "yVals" (innehållande hjärnvolym), använd ihåliga cirklar för att indikera punkter och anslutas med streckade linjer
- Titla tomten som: patientName Brain Volume Data
- Märk x -axeln enligt bilden
- Märk y -axeln enligt bilden
- Låt figur 5 vara lika med output
Från detta kommer funktionen patientnamn som anropas att ge en fil med redigerade data som håller koll på hjärnvolymen över tid och en plot som visar trender.
Steg 13: Stänga luckor i delplaner: Subplotclose.m

Funktionen, anpassad från kod från https://www.briandalessandro.com, fungerar för att stänga luckorna mellan huvudplottens siffror när figurerna som visar MR -bilderna och hjärnlagren skapas. Delplotfunktionen som används inom subplotclose.m justerar positionen för de givna delplanerna för att passa tätt mot varandra i aspekten av den längre dimensionen. Till exempel, om koden avser en 7 x 3 -matris, passar raderna tätt eftersom raddimensionen är längre. Om koden avser en 3 x 7 matris, passar kolumnerna tätt, med luckor i raderna, som visas i figurerna i vår huvudkod.
Steg 14: Huvudkoden: Rensa allt och be om inmatningar

För att starta huvudkoden, klicka på samma knapp som säger "Nytt" i det övre vänstra hörnet av fönstret och välj "Skript" istället för "Funktion" från de tidigare avsnitten. Skriv koden som visas på bilden i redigeringsfönstret. Kodraderna utför följande uppgifter i ordning:
- Stäng alla öppna filer utom 0, 1 och 2.
- Stäng alla figurfönster.
- Rensa alla variabler i arbetsytan.
- Rensa kommandofönstret.
- Visning i kommandofönstret: Ange följande dimensioner för MR -skanningarna:
- På en ny rad i kommandofönstret, fråga: Längd på en skanning i milimeter:. Svaret från användaren kommer att ställas in på variabeln "lengthMM".
- På en ny rad, fråga: Avståndet mellan MR -skanningar i millimeter:. Svaret från användaren kommer att ställas in på variabeln "ZStacks".
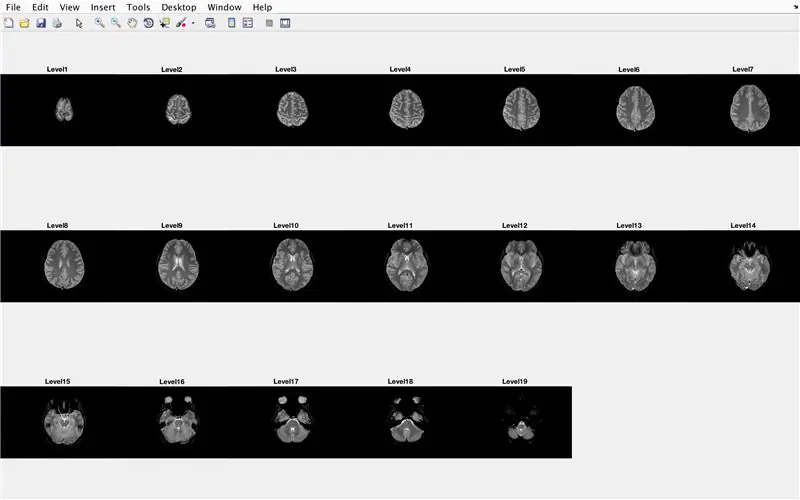
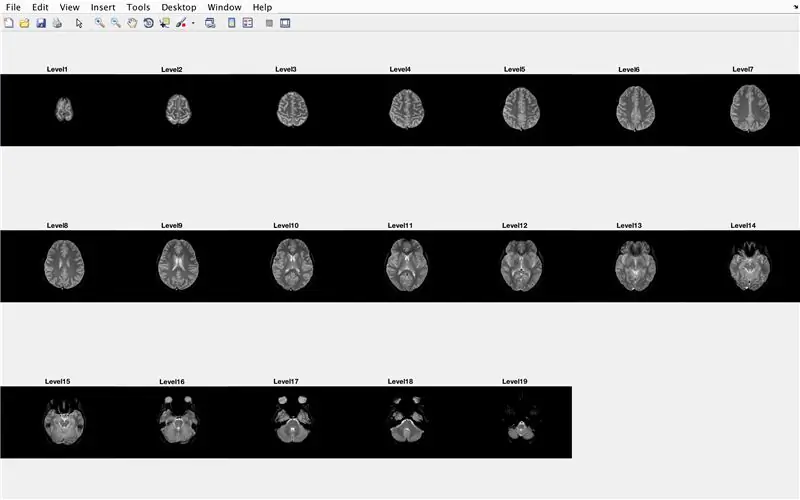
Steg 15: Huvudkoden: Batchbehandling av bilderna

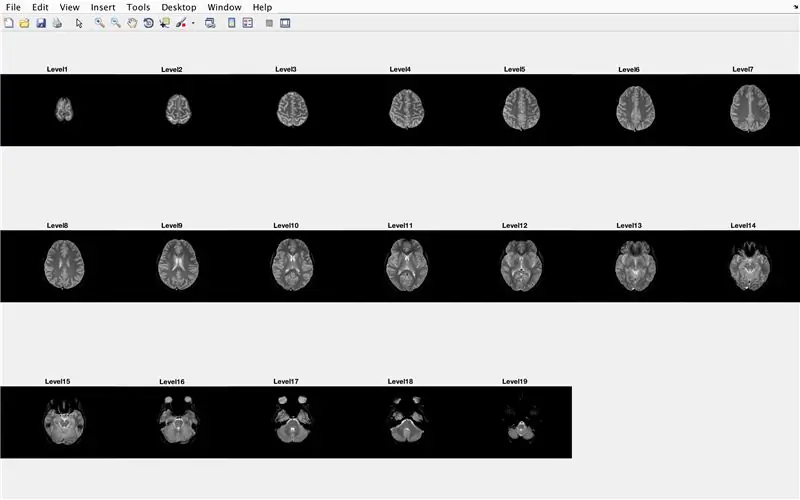
I detta avsnitt kommer koden att ladda upp bilderna (bestående av MR -skanningar av hjärnans tvärsnitt) och lagra namnen på varje bildfil i variabeln "Base" och visa var och en av MR -skanningarna. Följ koden på bilden, som gör följande:
- Skapa strukturmatris "BrainImages" som innehåller information om alla filer i den aktuella mappen som passar namnformatet för MRI _ (). Png
- Ange variabel "NumberofImages" lika med antalet element i strukturmatrisen "BrainImages"
- Öppna figurfönster 1
- Ställ in en for loop för att bläddra igenom för antalet bilder som räknas i filen
- För varje slinga är "CurrentImage" respektive namn på varje fil MRI_i.png, med iterationsnumret som 'i'
- Generera en 3 x 7 delplan för att visa de 19 bilder som ska laddas med "imshow"
- Visa varje bild som ett annat element i fönstret för delplottfigur
- Benämna varje delplotelement som Level_, där tomt är iterationsnumret för for -slingan.
- Avsluta for -loop (undvik fel)
Detta kommer att visa i figurfönster 1 alla MR -skanningar i rå form i en 3 x 7 -konfiguration utan luckor i x -orienteringen.
Steg 16: Huvudkoden: Padding

Med vaddering undviker vi problemet med små avvikelser i bildstorlekarna som kan ge fel vid dimensionerande fel om en bild är något större än en annan.
- Öppna figurfönster 2
- Ladda bildmatrisen från MRI_1-p.webp" />
- Hitta storleken på bildens matris och ställ in "OriginalXPixels" (för antal rader) och "OriginalYPixels" (för antal kolumner)
- Ställ in matrisen "BrainMat" så att den består av alla nollor med ytterligare 20 rader och ytterligare 20 kolumner för varje plan och 19 totala tvärsnitt, en per plan.
- Ställ in "HolesMat" för att bestå av samma tredimensionella uppsättning nollor för att mata in hålkoordinater senare
- Skapa "zeroMat" till storleken på plattan plus tjugo rader och tjugo kolumner, en tvådimensionell uppsättning nollor.
Steg 17: Huvudkoden: Fastställande av gränser
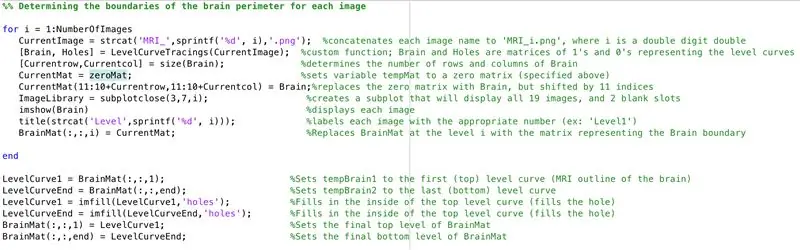
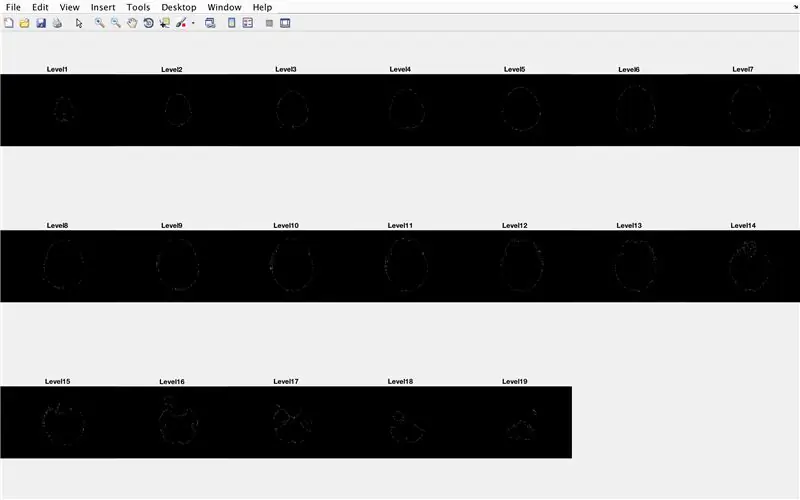
- Ställ in en for loop för att gå igenom data från varje bild som laddades tidigare
- På samma sätt för batchbehandling tidigare laddar "CurrentImage" filer med "MRI_i.png", där i är iterationsnumret
- Kör varje bild genom bearbetningsfunktionen "LevelCurveTracings2.m" som du gjorde tidigare
- Hitta storleken på utgången "Brain", ställ in antalet rader till "Currentrow" och antalet kolumner till "Current columns"
- Ställ in "CurrentMat" till en matris med nollor med de dimensioner som anges av "Currentrow" och "Currentcolumns"
- Centrera data från "Brain" i "CurrentMat", med en marginal på 10 rader på alla sidor
- Generera en delplan med måtten 3 x 7, för att visa gränserna för bilderna
- Titelera vart och ett av delplotelementen i figurfönstret
- Generera tredimensionell matris "BrainMat" som består av varje gränslager "CurrentMat"
- Avsluta for -loop (för att undvika fel)
Delen nedan fyller i hålen kvar på toppen och botten av den föreslagna tredimensionella formen
- Ställ in "LevelCurve1" lika med det första lagret av "BrainMat" (botten av solid)
- Ställ in "LevelCurveEnd" lika med det sista lagret av "BrainMat" (toppen av solid)
- Skriv över "LevelCurve1" med ett ifyllt lager
- Skriv över "LevelCurveEnd" med ett fyllt lager
- Ställ in det ifyllda lagret som det nedre lagret i "BrainMat"
- Ange det ifyllda lagret som det översta lagret av "BrainMat"
Steg 18: Huvudkoden: Bestämning av korrekt Z -dimension

De tre första raderna består av att ställa in en tom array "z" och göra enkla konverteringsoperationer (dela pixlar med längd) för att få en korrekt avläsning av volymen i mm^3
- Skapa en for -loop för att bläddra igenom varje lager
- Hitta antalet enor i ett visst lager
- Konvertera z -koordinaterna för dem till värden som skalas till rätt förhållande, inställt på "tempz", en kolumnvektor
- Lägg till z -värde för nivåkurvan till vektorn z
Med detta justeras z -koordinaterna ordentligt.
Steg 19: Huvudkoden: Bestämning av X- och Y -koordinater

Nu för att bestämma x- och y -positionerna för var och en av punkterna i gränserna.
- Initiera "xBrain" som en tom array
- Initiera "yBrain" som en tom array
- Ställ in en for loop för att gå igenom varje bild som laddas
- Kompilera en tvåkolumnmatris för att lagra de plana koordinaterna för varje punkt i gränsen, representerad av kolumnvektorerna "RowBrain" och "ColumnBrain"
- Lägg till "xBrain" med de nuvarande "RowBrain" -koordinaterna
- Lägg till "yBrain" med de nuvarande "ColumnBrain" -koordinaterna
- Avsluta for -slingan (för att undvika fel)
Steg 20: Huvudkoden: Plotta en tredimensionell struktur, hitta volym och logga data
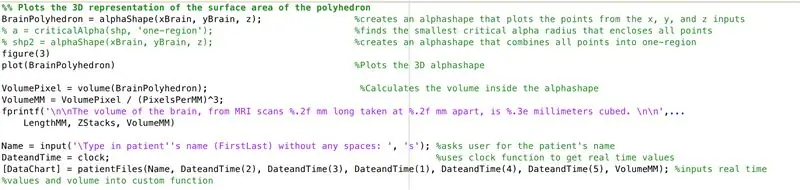

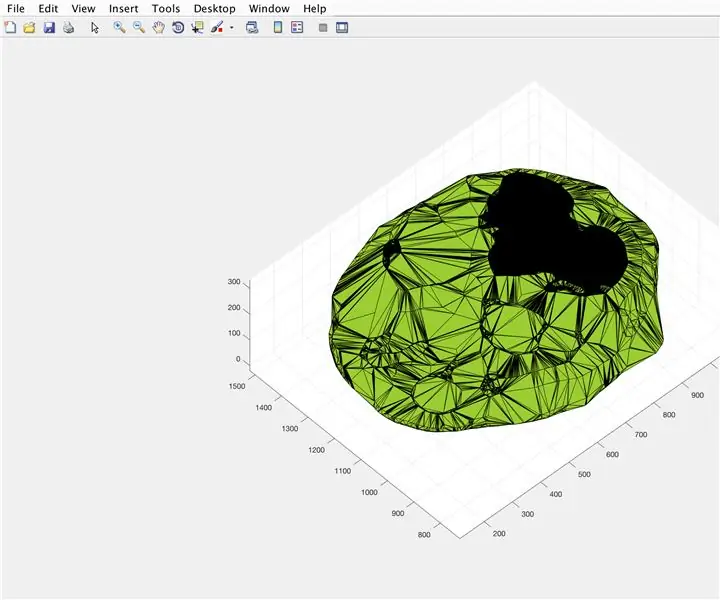
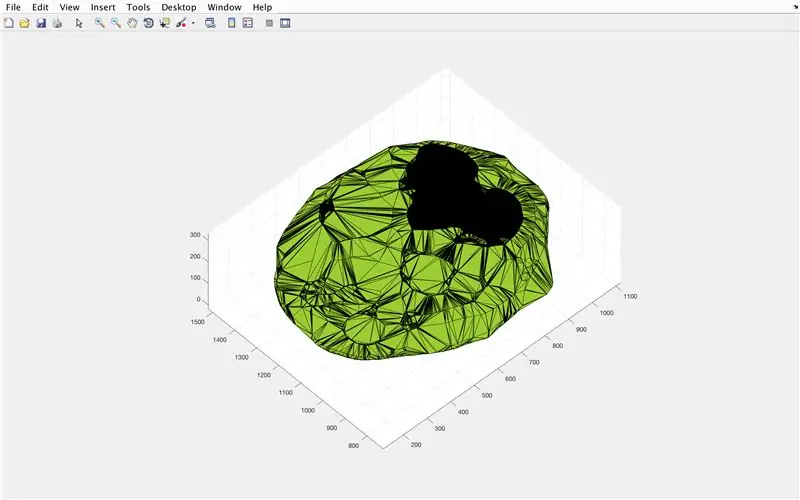
Med funktionen alphaShape kommer vi att skapa en tredimensionell struktur från vilken vi kan beräkna hjärnans volym.
- Använd funktionen alphaShape, koppla in vektorerna "xBrain", "yBrain" och "z" för x-, y- och z -koordinaterna och ställ in lika med "BrainPolyhedron"
- Öppna figurfönster 3
- Plotta den beräknade alfa -formen "BrainPolyhedron", visas i figurfönstret
- Beräkna volymen på alfa -formen med en funktion "volym" som fungerar för alfa -former
- Konvertera volymen till mm^3
- Skriv ut volymen för det fasta i kommandofönstret
- Fråga om att ett patientnamn ska anges som en ingång
- Få aktuellt datum och tid med klockan och ställ in på "DateandTime"
- Anropsfunktionen "patientFiles" för att logga och plotta de beräknade data
Härifrån ska den andra och tredje bilden visa siffrorna som dyker upp, och den fjärde bilden den som ska visas i kommandofönstret.
Rekommenderad:
Smart ryggsäck med GPS -spårning och automatiska ljus: 15 steg

Smart ryggsäck med GPS -spårning och automatiska lampor: I denna instruerbara gör vi en smart ryggsäck som kan spåra vår position, hastighet och har automatiserade lampor som kan hålla oss säkra på natten. Jag använder två sensorer för att upptäcka om det ligger på dina axlar för att se till att det inte släcks när det inte behöver,
Kvinnors säkerhetsenhet med GPS -spårning och varningar med Arduino: 6 steg

Kvinnors säkerhetsenhet med GPS -spårning och varningar med Arduino: Med all teknik tillgänglig för oss på senare tid är det inte svårt att bygga en säkerhetsanordning för kvinnor som inte bara genererar ett nödlarm utan också skickar ett meddelande till dina vänner, familj eller berörd person. Här bygger vi ett band
Volym, bas och diskantkrets i ljudförstärkare: 11 steg

Volym, bas och diskantkrets i ljudförstärkare: Hej vän, idag ska jag göra en krets med volym, bas och diskant.Denna krets kommer att styra förstärkarens och basens volym och den kommer också att styra förstärkarens diskant. kretsen kommer bara att vara för enkanals ljudförstärkare
Bas-, diskant- och volym -USB -kontroller med tangentbordets lysdioder: 6 steg (med bilder)
Bas-, diskant- och volym -USB -kontroller med tangentbordslampor: Jag har en Creative SoundBlaster Audigy i min stationära dator och jag behövde ett sätt att snabbt justera bas- och diskantinställningarna (samt volymen) när jag lyssnade på ljud- eller videomedier . Jag har anpassat koden från två källor i
PC -HÖGTALARE: Fjärrkontroll med trådbunden volym med parfymlock: 19 steg

PC -HÖGTALARE: Fjärrkontroll med trådbunden volym med parfymkåpa: (Före allt: förlåt mitt engelska jag från Brasilien …) Hej, min mamma köpte en edifier 2.1 PC -högtalare med trådbunden fjärrkontroll. (Foto 1) Det är väldigt trevligt och lätt att använda … men dyrt, och rattvolymen är lite liten … Jag gillar stora volymknappar, som
