Innehållsförteckning:
- Steg 1: Förkunskaper
- Steg 2: Förkunskaper (fortsätt)
- Steg 3: Förkunskaper (fortsätt)
- Steg 4: Förkunskaper (fortsätt)
- Steg 5: Rensa upp Matlab för att förbereda för körningskod
- Steg 6: Välj 10 normala ögonbilder och 10 bilder med diabetisk retinopati
- Steg 7: Välj 10 normala ögonbilder och 10 bilder med diabetesretinopati -symtom (fortsätt)
- Steg 8: Skapa 2 variabler (normala och diagnostiserade) och ställ dem var och en till 0
- Steg 9: Skapa en for Loop för att automatiskt ladda upp normala bilder
- Steg 10: Skapa en för loop för att automatiskt ladda upp normala bilder (fortsätt)
- Steg 11: Beskär bildens ramar
- Steg 12: Skapa en gråskalebild
- Steg 13: Skapa en kontrastbild
- Steg 14: Förbättra kontrastbilden
- Steg 15: Skapa ett genomsnittligt filter
- Steg 16: Kombinera medelvärdesfiltret med den kontrasterade bilden
- Steg 17: Gör en ny genomsnittsmask genom att subtrahera pixlar
- Steg 18: Skapa en binärfiltrerad bild
- Steg 19: Ta bort mindre klossar som finns i de filtrerade bilderna
- Steg 20: Skapa ett diskstruktureringselement
- Steg 21: Utför morfologiska stängningsoperationer
- Steg 22: Hitta objekt med anslutning av minst 8
- Steg 23: Hitta det maximala antalet anslutna pixlar
- Steg 24: Ställ in maxpixelvärdena på 0 och hitta pixlar med> = 26 pixelanslutningar
- Steg 25: Ta bort blodkärl i bilden
- Steg 26: Figurvisning
- Steg 27: Ta bort kärl och räkna blodkroppar
- Steg 28: Diagnostisera retinalbilden baserat på antalet identifierade blodproppar
- Steg 29: Om det finns fler än 5 klossar …
- Steg 30: Upprepa filtreringsprocessen för normala bilder med bildsiffror som 2 och 3
- Steg 31: Upprepa hela processen för de diagnostiserade bilderna
- Steg 32: Statistisk analys
- Steg 33: Hitta konfidensintervall
- Författare John Day day@howwhatproduce.com.
- Public 2024-01-30 12:46.
- Senast ändrad 2025-01-23 15:11.

(Se kodöversikten ovan)
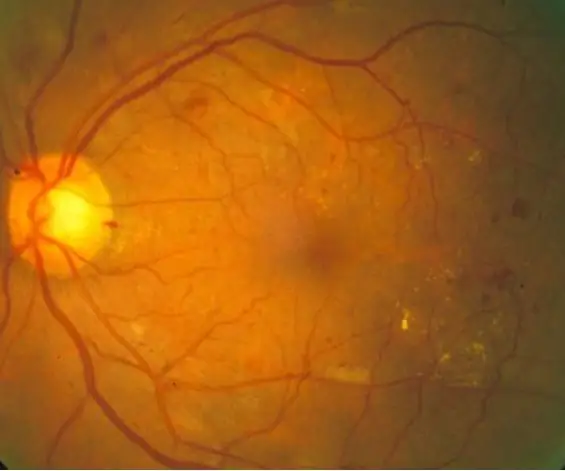
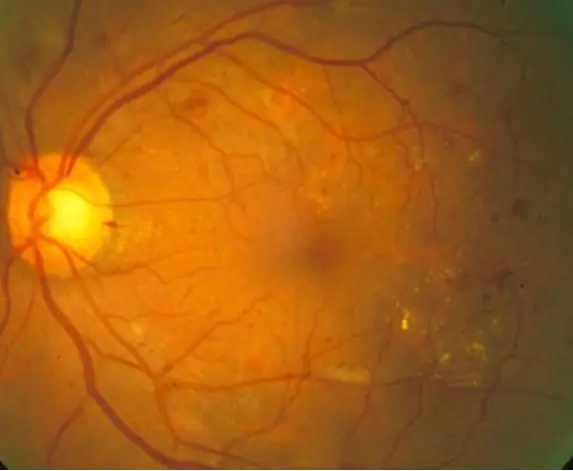
Diabetisk retinopati är en diabetesrelaterad ögonsjukdom som orsakas av höga blodsockernivåer. De höga blodsockernivåerna får blodkärlen i näthinnorna att svälla, vilket leder till förstorade blodkärl och till och med kärlläckor, vilket leder till mörka fläckar i näthinnebilder. Med denna kod syftar vi till att använda utseendet på blodkärlens läckfläckar som en indikator på diabetisk retinopati i bakgrunden, även om ytterligare diagnosmetoder skulle krävas i den verkliga världen. Målet med denna kod är att automatisera bildbehandling och diagnos av näthinnebilder för att identifiera tecken på diabetisk retinopati som visas genom mörka fläckar i näthinnebilderna.
10 normala näthinnebilder och 10 diagnostiserade näthinnebilder bearbetades genom en kod som först läser och filtrerar bilderna och sedan kvantifierar de mörka fläckarna för att avgöra om diabetiska retinopatisymtom finns, baserat på en given tröskel. Resultaten skrivs sedan ut i kommandofönstret för visningstolkning.
Steg 1: Förkunskaper
1. Se till att du har laddat ner MATLAB -programmet till din dator.
2. Ladda ner txt -filen som finns i länken. (Tryck på ‘ctrl+s’ för att spara i samma katalog som MATLAB -koden)
Steg 2: Förkunskaper (fortsätt)
4. Öppna MATLAB och skriv ‘uiimport’ i kommandofönstret.
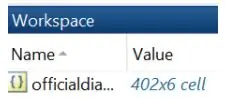
5. Välj filen officialdiagnoses.txt och importera den till MATLAB som en cellmatris.
6. Se till att du ser ‘officiell diagnos’ som en variabel i arbetsytan.
Steg 3: Förkunskaper (fortsätt)

7. Ladda ner funktionen ModWald.m, som kan hämtas från ovanstående kod eller ladda ner den från Canvas.
(Kod från professor King och professor Choi)
Steg 4: Förkunskaper (fortsätt)

8. Ladda ner de 400 råbilderna från datasektionen i STARE -projektet.
Steg 5: Rensa upp Matlab för att förbereda för körningskod

Lägg till i koden:
1. Stäng alla (Stänger alla tidigare öppnade bilder)
2. clearvars - utom officiell diagnos (rensar alla variabler utom den officiella diagnosen txt -filen som tidigare importerats)
3. cclc (rensar kommandofönstret)
Steg 6: Välj 10 normala ögonbilder och 10 bilder med diabetisk retinopati
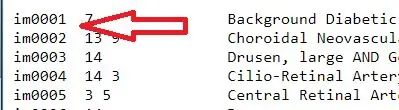
1. Ta diagnostextfilen och extrahera bildnamnen. Dessa namn finns i den första kolumnen i textfilen så för att extrahera dem skriver du 'officialdiagnoses (:, 1)'. Matrisen med bildnamn tilldelades en variabel "all_image_numbers"
2. Konvertera variabeln all_image_numbers från en cellmatris till en matrismatris med cell2mat -funktionen
Steg 7: Välj 10 normala ögonbilder och 10 bilder med diabetesretinopati -symtom (fortsätt)
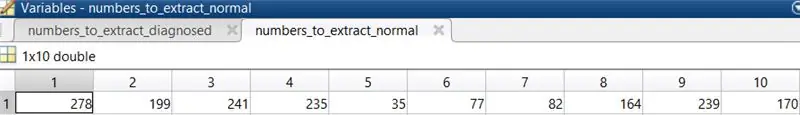
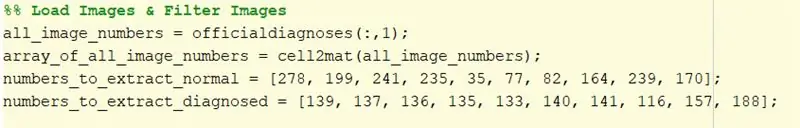
3. Välj 10 normala ögonbilder för att köra koden. Bilderna som valdes i detta fall var 278, 199, 241, 235, 35, 77, 82, 164, 239, 170.
Placera dessa nummer i en matris och tilldela dem till en variabel som kommer att kallas när bilderna laddas.
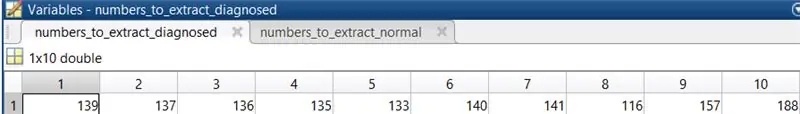
4. Upprepa steg 3 för näthinnebilder som har diagnostiserats med diabetisk retinopati. Bilderna som valdes i detta fall var 139, 137, 136, 135, 133, 140, 141, 116, 157, 188.
Steg 8: Skapa 2 variabler (normala och diagnostiserade) och ställ dem var och en till 0

Skapa dessa variabler före for -slingan för att initiera loopnumren.
Steg 9: Skapa en for Loop för att automatiskt ladda upp normala bilder

1. Skapa en for loop
2. Ställ in en räknarvariabel (i, i detta fall) till en matris med värden 1-10. Denna räkningsvariabel kommer att användas för att anropa varje bild individuellt
3. Ta i -elementet i bildmatrisen för att extrahera och konvertera bildnamnet från en sträng till ett tal med hjälp av num2str -funktionen.
Hitta antalet siffror som finns i bildnamnet med hjälp av numelfunktionen. Tilldela detta värde till en variabel, digits_normal. Detta nummer bör vara 1 för enkelsiffriga nummer, 2 för tvåsiffriga nummer och 3 för tresiffriga nummer. Denna information kommer att användas för att automatiskt ringa upp bilder.
Steg 10: Skapa en för loop för att automatiskt ladda upp normala bilder (fortsätt)

3. Skapa en if -sats som innehåller alla tre möjligheterna från föregående steg. Om bildnamnet har en siffra kallas bilden som”im000”, om den har två siffror kallas bilden som”im00”, och om den har tre kommer bilden att kallas som”im0”.
4. Under varje if -sats, tilldela en variabel för att läsa in "im" under motsvarande, if -sats med lämpligt antal nollor (enligt beskrivningen ovan), följt av i.
Steg 11: Beskär bildens ramar
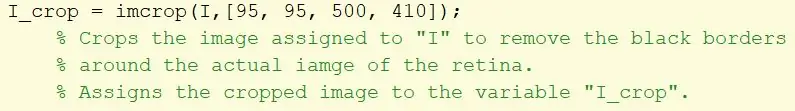
Ta den ursprungliga bilden och applicera ett imcrop -filter för att eliminera svarta kanter och tilldela en variabel I_crop. Skördarektangeln specificeras med hjälp av en matris [95, 95, 500, 410].
Steg 12: Skapa en gråskalebild

Ta den beskurna bilden och applicera rbg2gray -filtret för att ändra bilden till gråskala. Tilldela denna bild till variabel I2.
Steg 13: Skapa en kontrastbild
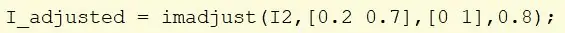
Ta bilden I2 och använd imadjust för att skala om intensitetsvärdena.
Ta värden som ligger inom intervallet [0,2, 0,7] och skala om dem till en [0, 1]. Gamma är inställd på 0,8 för att göra bilden ljusare. Tilldela den nya bilden till I_adjusted.
Steg 14: Förbättra kontrastbilden
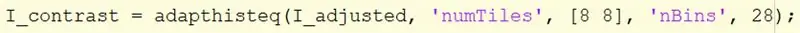
Ta den I_justerade bilden och använd adapthisteq -funktionen för att förbättra kontrasten.
Adapthisteq -syntaxen kräver bildnamnet, I_adjusted, ‘numTiles’, storleken på numTiles, ‘nBins’ och antalet fack. Storleken på numTiles är inställd på [8 8], uppdelning av bilden i 8x8 brickor och antalet fack är satt till 28. Tilldela bilden till I_constrast.
Steg 15: Skapa ett genomsnittligt filter

Skapa en variabel med namnet 'meanfilt' med fspecial -funktionen. Ange "genomsnittlig funktion" för att skapa medelvärdesfiltret och sätt in [90 90] för glidfönstret.
Steg 16: Kombinera medelvärdesfiltret med den kontrasterade bilden
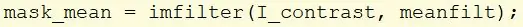
Skapa en ny variabel med namnet mask_mean och använd imfilter -funktionen för att ta I_contrast -bilden och tillämpa det genomsnittliga filtret som tidigare skapats.
Steg 17: Gör en ny genomsnittsmask genom att subtrahera pixlar

Skapa en variabel som heter mask_mean2 och använd funktionen imsubtract för att subtrahera värdet för varje pixel i I_contrast från motsvarande pixel i mask_mean.
Steg 18: Skapa en binärfiltrerad bild

Vänd gråskala bilder till svartvitt med hjälp av imbinarize. Mata in mask_mean2, 'adaptiv', 'ForegroundPolarity', 'dark', 'Sensitivity', 0.6. Tilldela denna nya bild mask_binarize.
Steg 19: Ta bort mindre klossar som finns i de filtrerade bilderna
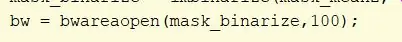
Ta bort objekt med anslutning mindre än 100 pixlar med bwareaopen -funktionen på mask_binarize och ställ in tröskelvärdet till 100. Tilldela variabeln som bw.
Steg 20: Skapa ett diskstruktureringselement

Skapa ett diskstruktureringselement (med en radie av 2) med hjälp av strel -funktionen. Tilldela det till se.
Steg 21: Utför morfologiska stängningsoperationer

Ta bw och applicera imclose -funktionen på strukturelementet för att utföra en morfologisk nära operation på objektet.
Steg 22: Hitta objekt med anslutning av minst 8
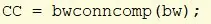
Ta bw och använd bwconncomp för att hitta objekt med en anslutning på minst 8 i bilden. Tilldela nummerutmatningen till cc_1.
Steg 23: Hitta det maximala antalet anslutna pixlar
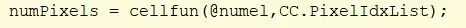
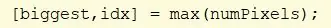
Använd cellfun -funktionen för att utföra funktionen "numel" på varje cell i CC. Detta hittar antalet element i PixelIdxList -cellen. Tilldela värde till “numPixels”.
Hitta maxvärdena i numPIxels. Tilldela det största max till “största” och indexet för maxvärdet till “idx”.
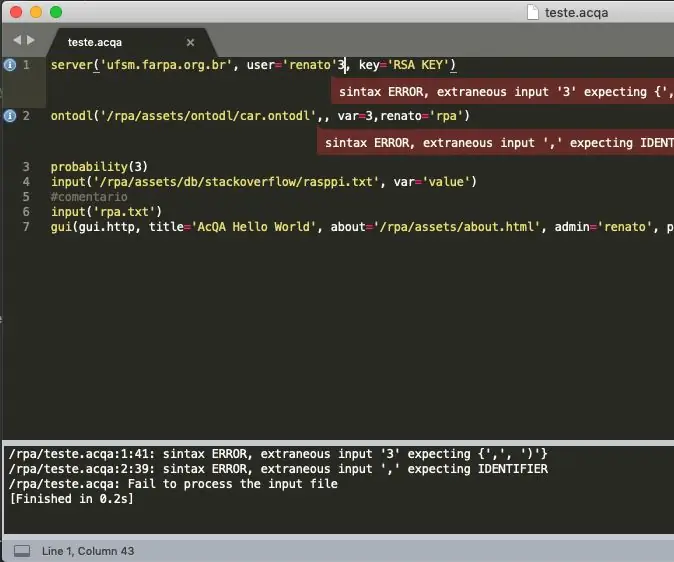
Steg 24: Ställ in maxpixelvärdena på 0 och hitta pixlar med> = 26 pixelanslutningar
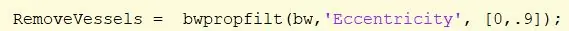
= 26 Pixel -anslutning "src =" https://content.instructables.com/ORIG/FXY/DTW3/JEOIIEL4/FXYDTW3JEOIIEL4-p.webp

= 26 Pixel -anslutning "src =" https://content.instructables.com/ORIG/FXO/GBX1/JEOIIELB/FXOGBX1JEOIIELB-p.webp

= 26 Pixel -anslutning "src =" {{file.large_url | add: 'auto = webp & frame = 1 & height = 300' %} ">

= 26 Pixel -anslutning "src =" {{file.large_url | add: 'auto = webp & frame = 1 & height = 300' %} ">
Ställ in pixlarna med de största värdena i bilden”bw” till 0, vilket gör pixlarna svarta.
Hitta objekten med en anslutning på minst 26 pixlar i bilden med hjälp av bwconncomp. Tilldela variabeln cc_1.
Steg 25: Ta bort blodkärl i bilden
Ta bort de blodkärl som fortfarande finns i bilden med bwpropfilt -funktionen med ett intervall på [0, 0,9].
[0,9, 1] utesluts eftersom värden nära 1 indikerar en rad. Tilldela “RemoveVessels”.
Steg 26: Figurvisning
Visa varje filtrerad bild i en delplan. Imshow. med "gräns" och "snäva" ingångar, visar varje bild i en delplotstruktur. Lägg till en titel till varje bild för att skilja vilket filter som användes.
Steg 27: Ta bort kärl och räkna blodkroppar
1. Ta “RemoveVessels” och använd funktionen”Centroid” i regionprops för att identifiera centroiderna för objekten i bilden. Dessa föremål ska motsvara blodproppar i bilden.
2. Räkna antalet blodproppar som identifierats genom att ta längden på centroidmatrisen.
Steg 28: Diagnostisera retinalbilden baserat på antalet identifierade blodproppar
Använd if -uttalanden för att diagnostisera bilden baserat på antalet identifierade blodproppar.
Om antalet centroider som identifierades var mindre än eller lika med 5, identifierades bilden som normal.
Om antalet centroider var större än 5, diagnostiserades bilden med diabetisk retinopati.
Resultatet skrivs ut på kommandofönstret med fprintf.
Steg 29: Om det finns fler än 5 klossar …
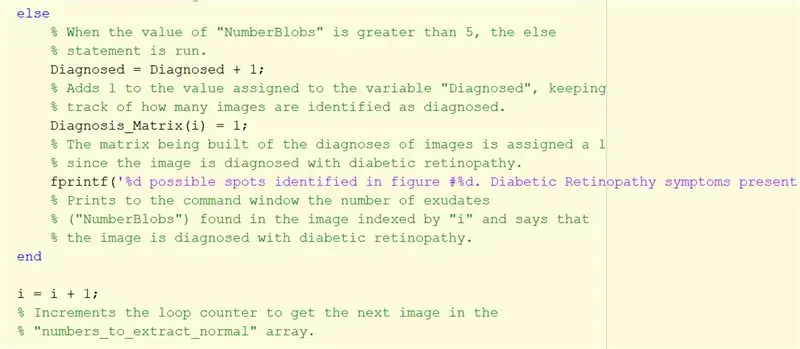
Upprepa instruktionerna ovan för diagnostiserade bilder som ett annat uttalande. Den här delen körs om antalet klossar är större än 5.
Avsluta if -uttalandet.
Steg 30: Upprepa filtreringsprocessen för normala bilder med bildsiffror som 2 och 3
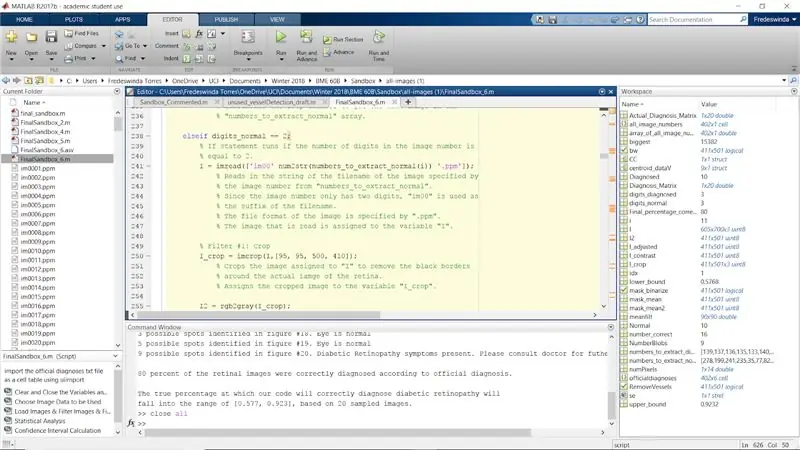
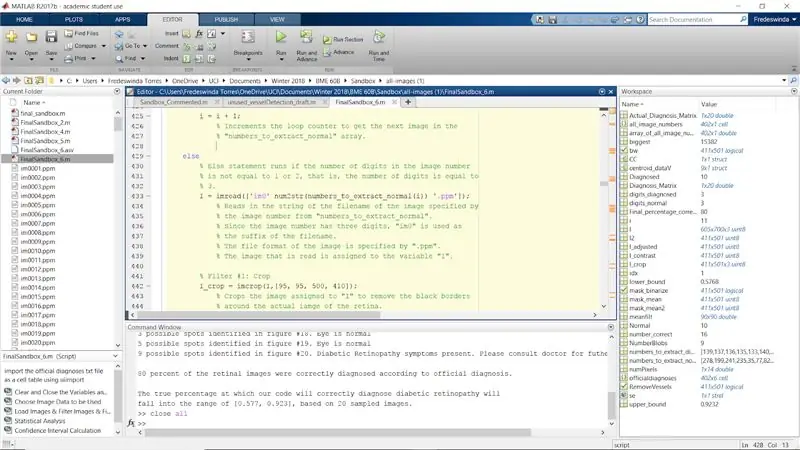
Upprepa processen för resten av originalet om påståenden när numel (antalet siffror i bildnumret) är lika med 2 och 3. Detta slutför för -slingan för de normala bilderna.
Avsluta for -slingan.
Steg 31: Upprepa hela processen för de diagnostiserade bilderna
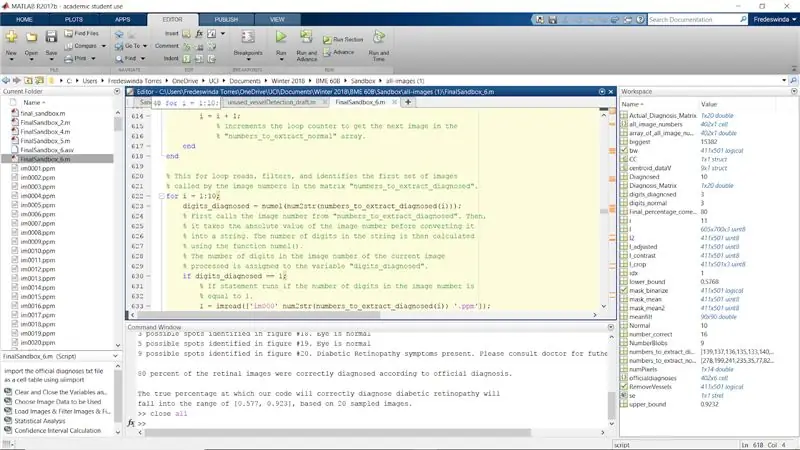
Upprepa hela processen med de diagnostiserade bilderna som listas av matrisen "numbers_to_extract_diagnosed".
Se till att gå igenom varje siffra (i) och ändra den till siffra (i+10) så att de diagnostiserade siffrorna dyker upp som bilder 11 till 20.
Steg 32: Statistisk analys
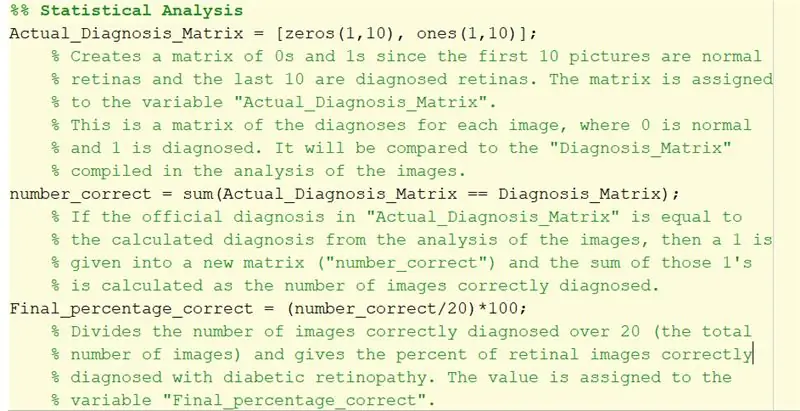
1. "Actual_Diagnosis_Matrix" används för att jämföra resultaten med den officiella diagnosen som finns i txt -filen. De första 10 nollorna indikerar att de första 10 bilderna ska vara normala. De tio sista indikerar att de tio sista bilderna ska klassificeras som diabetisk retinopati.
2. Det dubbla likhetstecknet som används för att skapa "number_correct" skapar en logisk array genom att jämföra värdet för motsvarande element i "Actual_Diagnosis_Matrix" med "Diagnosis_Matrix" som skapats från for -slingan.
För varje element som matchar diagnosen kommer ett 1 att läggas till, vilket betyder att koden korrekt diagnostiserade den bilden. Om den är felaktig kommer den att lägga till en 0 i matrisen.
Sedan tar summan av det sammanlagt alla. Med andra ord, den hittar summan av korrekt diagnostiserade bilder.
3. 'Final_percentage_correct' är den beräknade procentandelen av hur exakt koden diagnostiserade diabetisk retinopati. Antalet korrekt diagnostiserade bilder divideras med 20 (det totala antalet bilder) och multipliceras med 100 för att hitta andelen framgångsrika diagnoser.
Steg 33: Hitta konfidensintervall
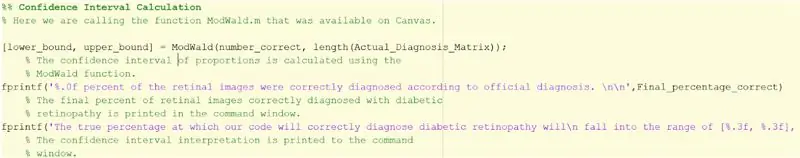
1. Se till att du har laddat ner ModWald.m för att kalla det som en funktion. Utan funktionen måste du själv beräkna konfidensintervallet med den modifierade Wald -metoden.
2. ModWald -funktionen har 2 ingångar där den första är antalet korrekt identifierade bilder och den andra är det totala antalet bilder.
3. ModWald -funktionen kommer att mata ut de nedre och övre gränserna för konfidensintervallet för proportioner för noggrannheten hos de samplade data. Med andra ord, det ger dig ett intervall av procentsatser där den verkliga procenten av kodens noggrannhet kommer att ligga.
4. Använd fprintf nedan för att mata ut statistik och konfidensintervall till kommandofönstret.
> fprintf ('%. 0f procent av näthinnebilderna diagnostiserades korrekt enligt officiell diagnos. / n / n', Final_percentage_correct)
> fprintf ('Den sanna procenten med vilken vår kod korrekt diagnostiserar diabetisk retinopati kommer / n att falla inom intervallet [%.3f, %.3f], baserat på 20 samplade bilder / n', lower_bound, upper_bound)
Rekommenderad:
Automatiserad EKG -kretsmodell: 4 steg
Automatiserad EKG -kretsmodell: Målet med detta projekt är att skapa en kretsmodell med flera komponenter som på ett adekvat sätt kan förstärka och filtrera en inkommande EKG -signal. Tre komponenter kommer att modelleras individuellt: en instrumentationsförstärkare, ett aktivt hackfilter och en
Automatiserad EKG -kretssimulator: 4 steg
Automatiserad EKG -kretssimulator: Ett elektrokardiogram (EKG) är en kraftfull teknik som används för att mäta den elektriska aktiviteten i en patients hjärta. Den unika formen på dessa elektriska potentialer skiljer sig beroende på plats för inspelningselektroder och har använts för att detektera många
Parkeringshjälp Enkel reparation / diagnos: 4 steg

Parkeringsassistent Enkel reparation / diagnos: Ok, kom igång, jag har en 2010 Chevrolet Avalanche och den har 4 parkeringsassistentsensorer i den bakre stötfångaren. Denna svårslipade kan användas på och fordon enligt min bästa vetskap, väder du har fram eller Rea eller båda. Så jag gick till min favorit
Hårddiskar: Diagnos, felsökning och underhåll: 3 steg

Hårddiskar: Diagnos, felsökning och underhåll: Vad är en hårddisk?- Helt enkelt är det hårddisken som lagrar all din data. Den rymmer hårddisken, där alla dina filer och mappar är fysiskt belägna. Informationen lagras magnetiskt på skivan, så den förblir på enheten även när
Inventering - Snabb PC -diagnos nu ersatt, se nedan: 6 steg

Inventering - Snabb PC -diagnos nu ersatt, se nedan: Om du någonsin behöver arbeta på en dator som inte ligger framför dig behöver du en noggrann inventering. Detta instruerbara uppstod bara för att en kollega har problem med att hitta en drivrutin till en dator. Jag kommer att använda Aida32 personal från http://majorgeeks.com
